Nat Med:基于22种常见癌症NGS数据开发AI模型,可对未知原发性癌症类型进行分类并预测治疗反应
2023-08-21 测序中国 测序中国 发表于上海
研究团队开发了一个名为OncoNPC的AI模型,并使用来自3家机构的22种常见癌症类型(共包括36,445个肿瘤)的NGS数据对其进行训练。
癌症可细分成许多类型,大多数情况下是按照发源的器官部位命名,与其在体内扩散的位置无关。现有癌症诊疗体系中,明确原发部位是进行标准化治疗的基础。但在3%-5%的癌症患者中,尤其是肿瘤已发生全身转移的患者,医生通过详细诊断检查无法确定癌症的原发部位,这些肿瘤被称为原发灶不明癌(CUP)。CUP原发部位隐匿、预后极差,中位生存期仅为6-16个月,是癌症死亡的重要原因之一。
新兴的肿瘤靶向疗法都是针对特定的癌症类型而研发的,由于原发部位不明,现有治疗方案不适用于CUP患者。因此,准确识别CUP肿瘤的潜在原发部位并证明位点特异性,可为CUP患者提供更多的治疗选择。与前瞻性试验相比,回顾性电子健康记录(EHR)数据可以捕捉到更大、更异质的患者群体。因此,将EHR数据与肿瘤测序相结合,可深入了解CUP以及其与患者预后的关系。
近日,美国丹娜-法伯癌症研究所、纪念斯隆-凯特琳癌症中心等机构的研究人员合作在Nature Medicine上发表了题为“Machine learning for genetics-based classification and treatment response prediction in cancer of unknown primary”的文章。研究团队开发了一个名为OncoNPC的AI模型,并使用来自3家机构的22种常见癌症类型(共包括36,445个肿瘤)的NGS数据对其进行训练。在65.2%的肿瘤样本,OncoNPC实现了了高置信度预测,加权F1值为0.942。OncoNPC不仅能确定CUP起源,还可区分CUP患者的预后,其使本可接受基因组指导治疗的CUP患者增加2.2倍,为CUP精准靶向治疗带来曙光。

文章发表于Nature Medicine
OncoNPC是一种基于多中心靶向测序数据训练的原发性癌症类型分类器(图1),其利用体细胞改变,包括突变(SNV和InDel)、突变特征、拷贝数改变(CNA)和患者测序时的年龄及性别,并通过XGBoost算法联合预测癌症类型。研究团队收集了36,445个原发和转移肿瘤样本对OncoNPC进行训练和验证,共涵盖22种已知癌症类型。结果显示,OncoNPC能准确地对22种不同类型的癌症进行分类。
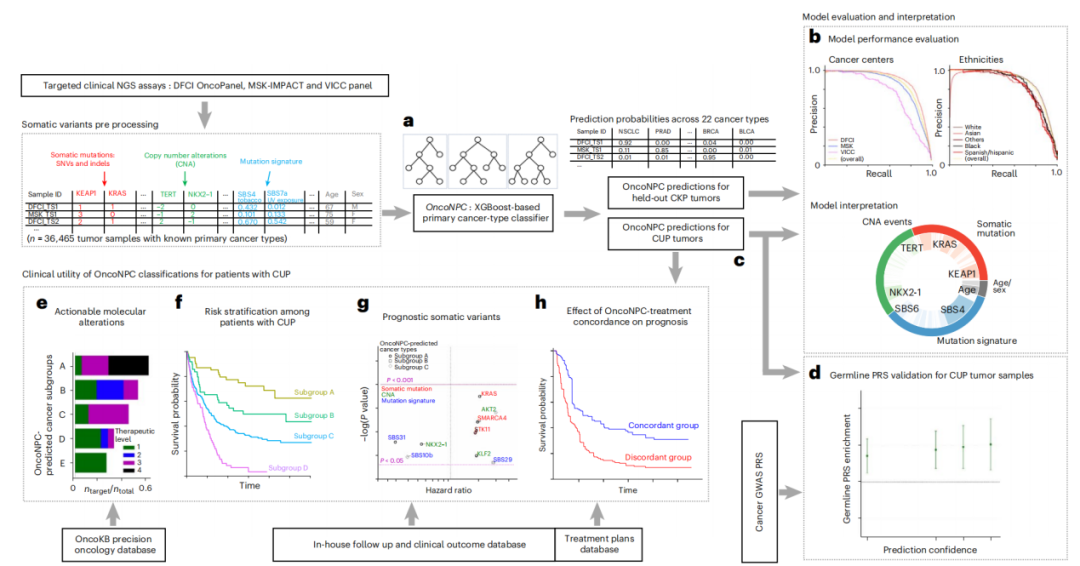
图1. 人工智能模型的开发和分析工作流程概述。
基于pmax(即22种癌症类型的最大预测概率),研究团队在0.0、0.5、0.7和0.9等四个不同的预测置信水平下评估了OncoNPC的性能(图2)。结果显示,基于pmax的阈值可进一步提高OncoNPC性能,增加pmax阈值可减少常见/罕见癌症类型之间的差异。此外,OncoNPC对由癌症中心、活检部位类型等因素导致的数据集变化表现出稳健的性能。
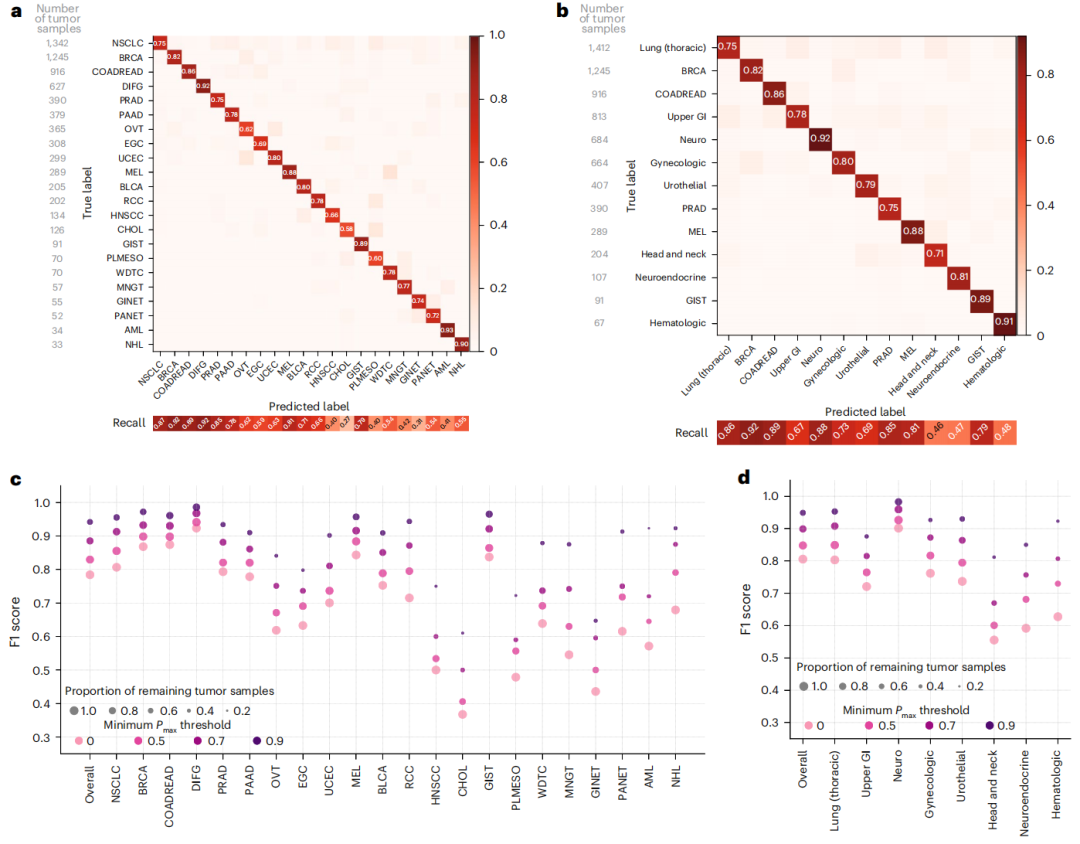
图2. OncoNPC可用于预测原发灶不明癌的起源。
研究团队通过OncoNPC对来自丹娜-法伯癌症研究所的971例CUP肿瘤进行分析(图3)。结果显示,对于41.2%的CUP肿瘤,该模型能够做出高置信度预测。其中,最常见的CUP癌症类型为非小细胞肺癌、胰腺癌、乳腺癌、早期胃癌和结直肠癌等,这些癌症发病率与先前研究结果基本一致。
接下来,研究团队探究了原发灶已知癌(CKP)队列和CUP队列中可预测每种癌症类型的最具影响力的特征,以评估OncoNPC的有效性。对于非小细胞肺癌, CKP和CUP肿瘤样本最重要的特征是EGFR突变和SBS4;对于乳腺癌,CKP和CUP肿瘤样本最重要的特征是性别。上述结果表明,OncoNPC能够在个体和队列水平上捕捉体细胞突变和临床特征中的癌症特异性信号。
研究团队假设,如果OncNPC可准确识别潜在的原发性癌症,CUP癌症的分类类型将显示出相应癌症的种系风险增加。为此,研究团队估算了每位CUP患者共同的种系变异,并使用癌症全基因组关联研究(GWAS)数据量化了8种常见癌症中的多基因风险评分(PRS)。结果显示,与其他癌症类型相比,CUP患者OncoNPC预测的PRS显著更高。
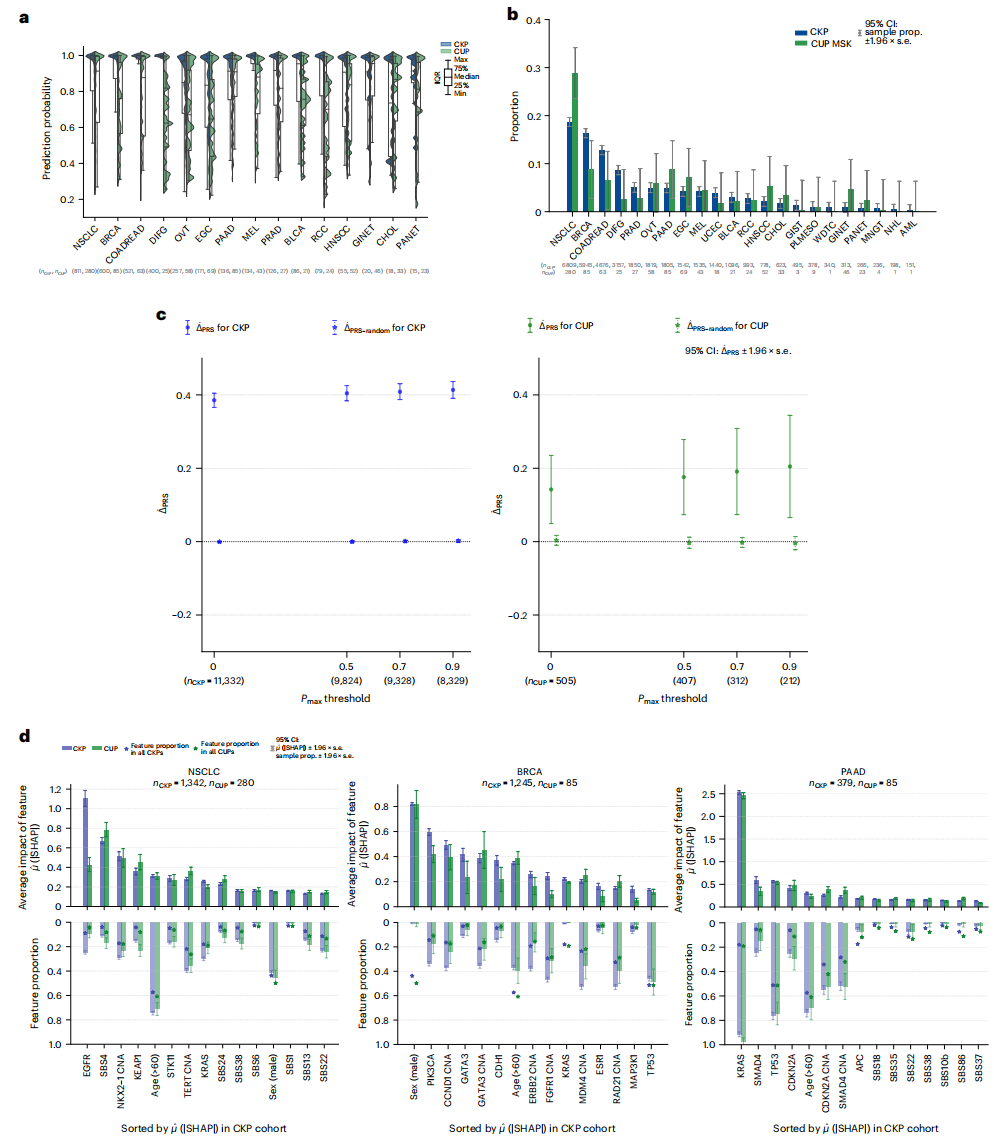
图3. OncoNPC在CUP肿瘤样本中的应用。
为证明OncoNPC的临床实用性,研究团队分析了具有中高置信度的OncoNPC癌症类型预测能否对CUP患者的总体生存率进行分层。根据OncoNPC预测,研究团队确定了生存率具有显著预后差异的亚组(图4)。总体而言,预测为早期胃癌和胰腺癌的CUP患者预后最差;预测为头颈部鳞癌的CUP患者中观察到最有利的预后。上述结果表明,OncoNPC可预测、捕获每种癌症类型的特异性预后信号,因此能够依据肿瘤OncoNPC亚组对CUP患者的生存率进行有意义的分层。
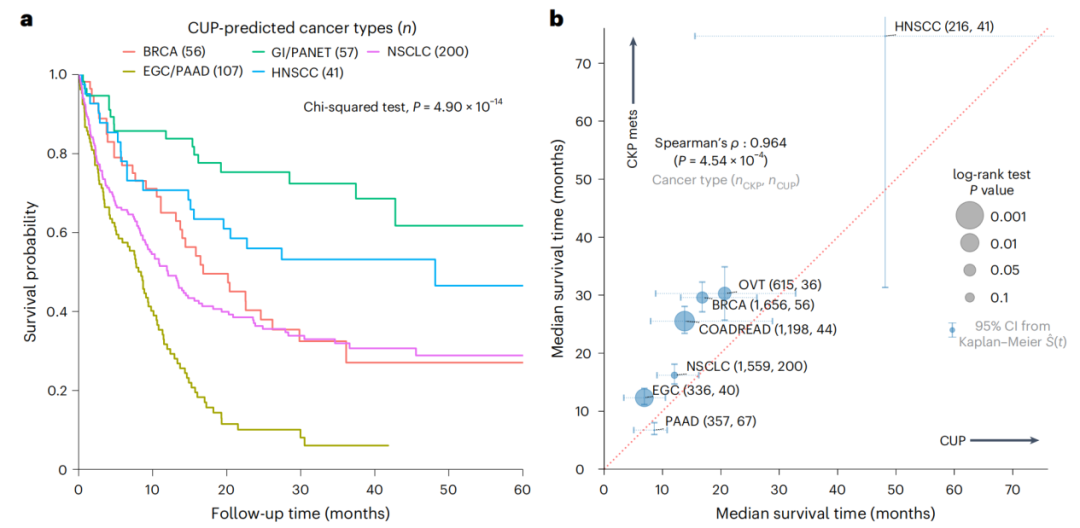
图4. CUP患者中基于肿瘤NPC的风险分层。
研究团队还进行了一项回顾性生存分析,以调查CUP患者在接受与OncoNPC预测一致的治疗时是否获得了临床益处(图5)。具体而言,共有158名CUP患者在丹娜-法伯癌症研究所接受第一次治疗,研究团队根据美国国家综合癌症网络(NCCN)指南,以确定所给予的治疗是否与OncoNPC预测一致。结果表明,首次接受治疗的CUP患者与OncoNPC预测的癌症类型一致,其生存率显著高于接受不一致治疗的患者。
根据158名CUP患者的EHR数据,研究团队确定了20名可接受基因组指导治疗的患者(12.7%),在这些患者中,依据OncoNPC预测肿瘤的方式接受治疗的CUP患者,其生存期明显长于以不一致方式治疗的患者。利用该模型,研究团队还确定了另外15%可接受基因组指导治疗的患者(增加2.2倍)。此外,将可操作的体细胞变异与预测的癌症类型相结合,研究团队得到28种符合1-3级条件的药物,其中1级对应于FDA批准的药物,2级对应于标准护理,3级对应于生物证据。
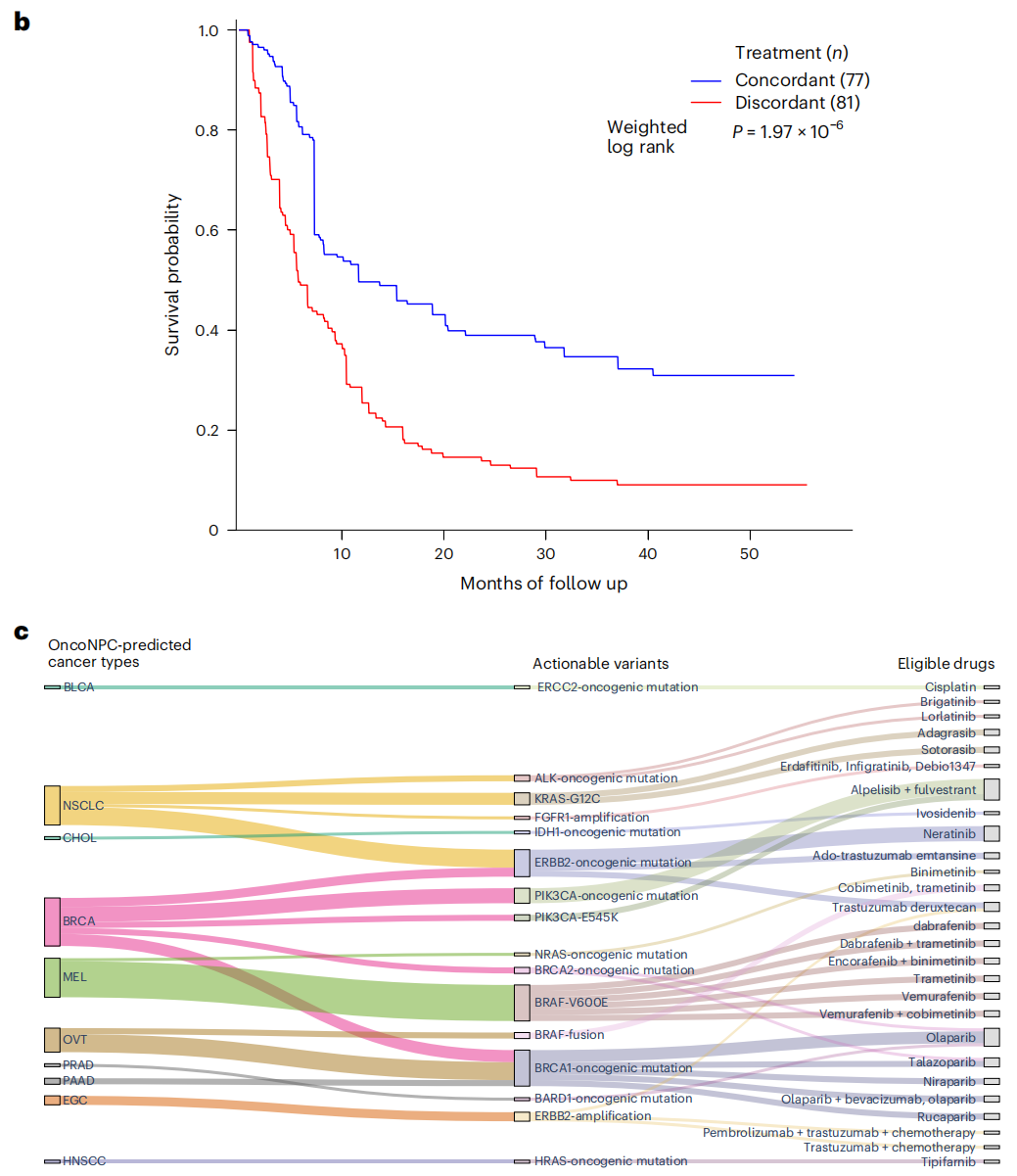
图5. 基于OncoNPC指导CUP患者的治疗决策。
综上所述,该研究开发了机器学习模型OncoNPC,可对肿瘤样本进行分类,并在已公布的多中心测试数据中提供了可靠且可解释的预测。研究团队观察到,通过OncoNPC预测方式进行治疗的CUP患者的生存期明显长于以不一致方式治疗的患者。OncoNPC极大地方便了个性化治疗的实施,对医生为CUP患者制定合适、精准的治疗策略具有重要意义。
参考文献:
Moon I, LoPiccolo J, Baca SC, Sholl LM, Kehl KL, Hassett MJ, Liu D, Schrag D, Gusev A. Machine learning for genetics-based classification and treatment response prediction in cancer of unknown primary. Nat Med. 2023 Aug 7. doi: 10.1038/s41591-023-02482-6. Epub ahead of print. PMID: 37550415.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言













