Nature:All of Us项目发表超24万个临床级基因组数据,鉴定超2.75亿种新变异
2024-02-29 测序中国 测序中国 发表于上海
该研究鉴定了超过10亿种遗传变异,包括超过2.75亿种新的遗传变异,其中超过390万种为编码变异。
2018年5月,由美国国立卫生研究院(NIH)领导的All of Us项目开放注册,其初步规划目标是建立一个由100万或更多美国人组成的大型研究队列,收集全面健康数据,以扩展精确医疗方法的知识平台。All of Us的一个重要组成部分是生成100万参与者的全基因组序列(WGS)和基因分型数据,并将这些数据公开供科学界使用,从而促进遗传祖先、遗传性疾病风险和药物遗传学等方面的研究。
近日,All of Us研究团队在国际顶尖期刊Nature发表了题为“Genomic data in the All of Us Research Program”的文章,报道了来自245388名All of Us参与者的临床级WGS数据,并展示了这种高质量数据在遗传和健康研究中的影响。该研究鉴定了超过10亿种遗传变异,包括超过2.75亿种新的遗传变异,其中超过390万种为编码变异。研究团队利用基因组数据和纵向电子健康记录之间的联系,评估了与117种疾病相关的3,724种遗传变异,发现欧洲血统和非洲血统的参与者都有很高的复制率。这些研究数据已上传到All of Us Researcher Workbench云平台,在保护参与者隐私的前提下促进公平的数据和计算访问,以此推动该领域的发展。

文章发表在Nature
主要研究内容
All of Us研究计划
目前,All of Us已有超过75万人参加,参与者数据包括表型和基因组数据的丰富组合,数据包括245388个WGS和312925个参与者的全基因组基因分型。本次发布的测序和基因分型数据没有基于任何临床或表型特征进行优先排序。值得注意的是,99%拥有WGS数据的参与者还拥有身体相关数据,84%拥有电子病历数据。在本次数据发布中,其中46%的人自我认同为少数民族群体。
从样本采集到测序,All of Us的基因组学工作都符合临床实验室标准,所有生物标本(包括血液和唾液)都被送到All of Us生物样本库进行处理和储存。All of Us数据和研究中心维护所有参与者信息和生物标本ID链接,以保护参与者的隐私。
为了满足临床对DNA样本提取和测序的准确性、精确性和一致性的要求,All of Us基因组中心建立了标准的质控方法和指标,并使用先前表征的临床样本和市售参考标准进行了一系列验证实验。总的来说,这些数据不仅具有临床级质量,而且在基因组中心的覆盖范围和一致性方面也保持一致。变异检测显示,单核苷酸变异(SNV)的总体敏感性大于98.7%,精密度大于99.9%。对于短插入或缺失(Indel),灵敏度大于97%,精密度大于99.6%。
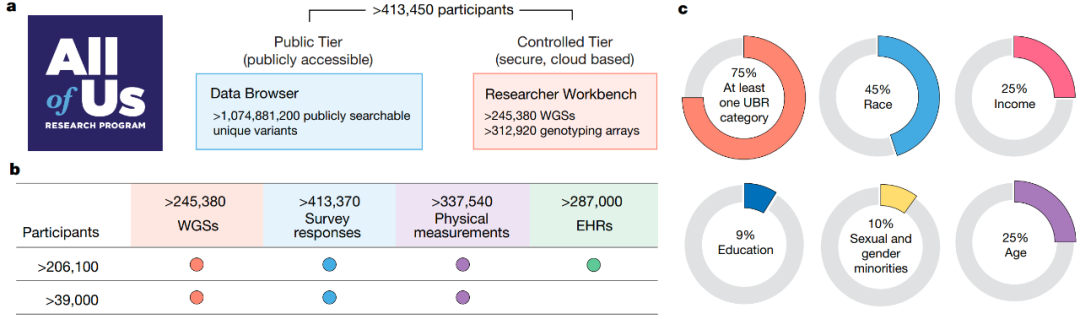
图1. All of Us数据资源概况
联合检测集包括超过10亿个基因变异,研究发现了272,051,104个非编码变异和3,913,722个编码变异,这些变异此前在dbSNP v153中没有描述过。共有3,912,832个(99.98%)编码变异是罕见的,其余883个是常见的。在编码变异中,454个在All of Us的一个或多个非欧洲祖先个体中常见,在欧洲祖先的参与者中罕见,并且等位基因数大于1000。
特别是,研究发现欧洲血统亚群的致病性变异率最高(2.1%),是东亚血统个体致病性变异率的2倍。东亚个体的变异频率较低,部分原因可能是该群体的样本量较小,而且变异数据库中可能存在信息偏差,从而减少了一些研究较少祖先群体的变异发现数量。
遗传祖先和亲缘关系
遗传祖先推断证实,51.1%的All of Us WGS数据集来自非欧洲血统的个体,研究人员在基于3202个样本和151159个常染色体单核苷酸多态性(SNP)的主成分分析空间上训练了一个分类器。基于来自WGS数据的相同SNP,研究人员将All of Us样本映射到训练数据的主成分分析空间中,并从训练的分类器中生成分类祖先预测。结果表明,使用全基因组基因型进行的祖先推断与真实数据高度一致。
亲缘关系评估层面的分析表明,约85%在数据集中没有一级或二级亲属,符合所有WGS数据主要由无血缘关系个体组成的事实。
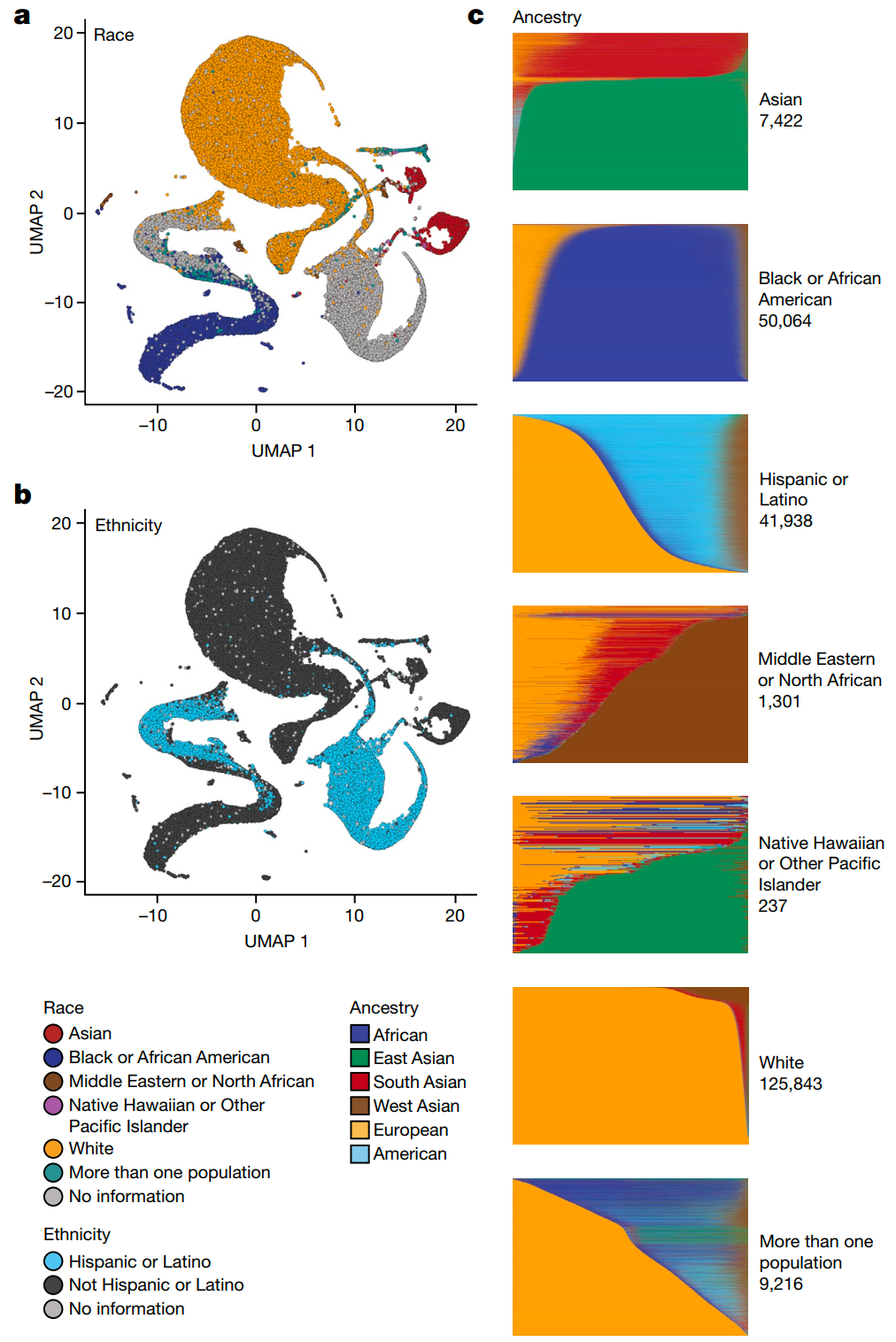
图2. All of Us中的遗传祖先分析
LDL-C的遗传决定因素
作为数据质量和效用的衡量标准,研究团队对低密度脂蛋白胆固醇(LDL-C)进行了单变异全基因组关联研究(GWAS),这是一种具有良好基因组结构的性状。在245388名WGS参与者中,91749人有一次或多次LDL-C检测。All of Us中LDL-C GWAS鉴定了20个完善的全基因组显著位点。
随后,研究人员将结果与近期发表的类似研究进行比较,发现两者的全基因组显著位点的效应估计具有很强的相关性。
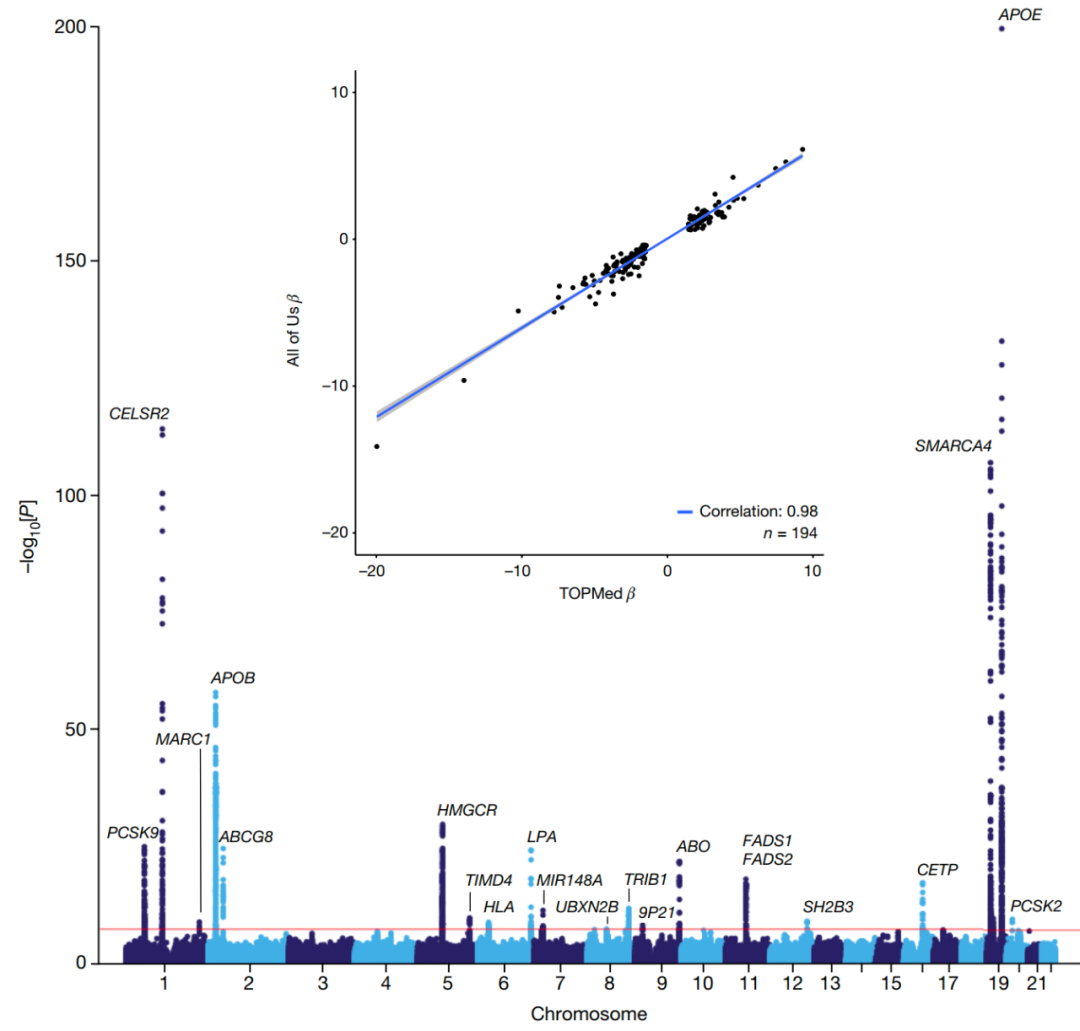
图3. All of Us中LDL-C的GWAS
基因型与表型的关联
研究人员还分析了已报道的表型-基因型关联在表型/基因型参考图谱(PGRM)中存在的五个预测遗传祖先群体中的重复率:AFR,非洲祖先;AMR,拉丁裔/混合美国血统;EAS,东亚血统;EUR,欧洲血统;SAS,南亚血统。
研究团队专门分析了4947个变异,计算了每个祖先群体中强大关联的复制率。80%关联的总体复制率为:AFR为72.0%,AMR为100%,EAS为46.6%,EUR为74.9%,SAS为100%。除了EAS祖先的结果外,这些高复制率与已发表的PGRM分析相当,其中几个单点EHR连接生物库的复制率在76%至85%之间。该分析证明了数据的实用性,也强调了进一步了解All of Us人群的特点和基因-环境相互作用对基因型-表型定位的潜在贡献。
例如,达菲血型位点(ACKR1)在AFR血统和AMR血统的个体中比在EUR血统的个体中更普遍。虽然对该位点的全表型关联研究强调了达菲血型与AFR和AMR祖先个体中白细胞计数较低的良好关联,也揭示了遗传-祖先特异性表型模式,其在EAS血统个体和EUR血统个体中具有最小的表型关联。
研究将数据上传到云平台,在获得访问权限后,研究者可以在任何时候创建一个新的工作空间来进行研究,前提是遵守所有的数据使用政策并声明自己的研究目的。对于已有数据使用协议的研究人员,只要完成所需的验证和合规步骤,就可以获得访问权限。
此外,鉴于该项目的表型和基因组数据集的规模在2023年达到4.75PB,与允许研究人员下载基因组数据的典型方法相比,使用中央数据存储和云分析工具每年将节省约1650万美元。在556所注册机构中为每个机构存储一份这些数据的副本每年将花费约11.6亿美元,相比之下,存储一个中央云副本每年的成本约为114万美元,节省了99.9%。重要的是,云平台设施还使数据访问民主化,特别是对于没有高性能本地计算资源的研究人员。
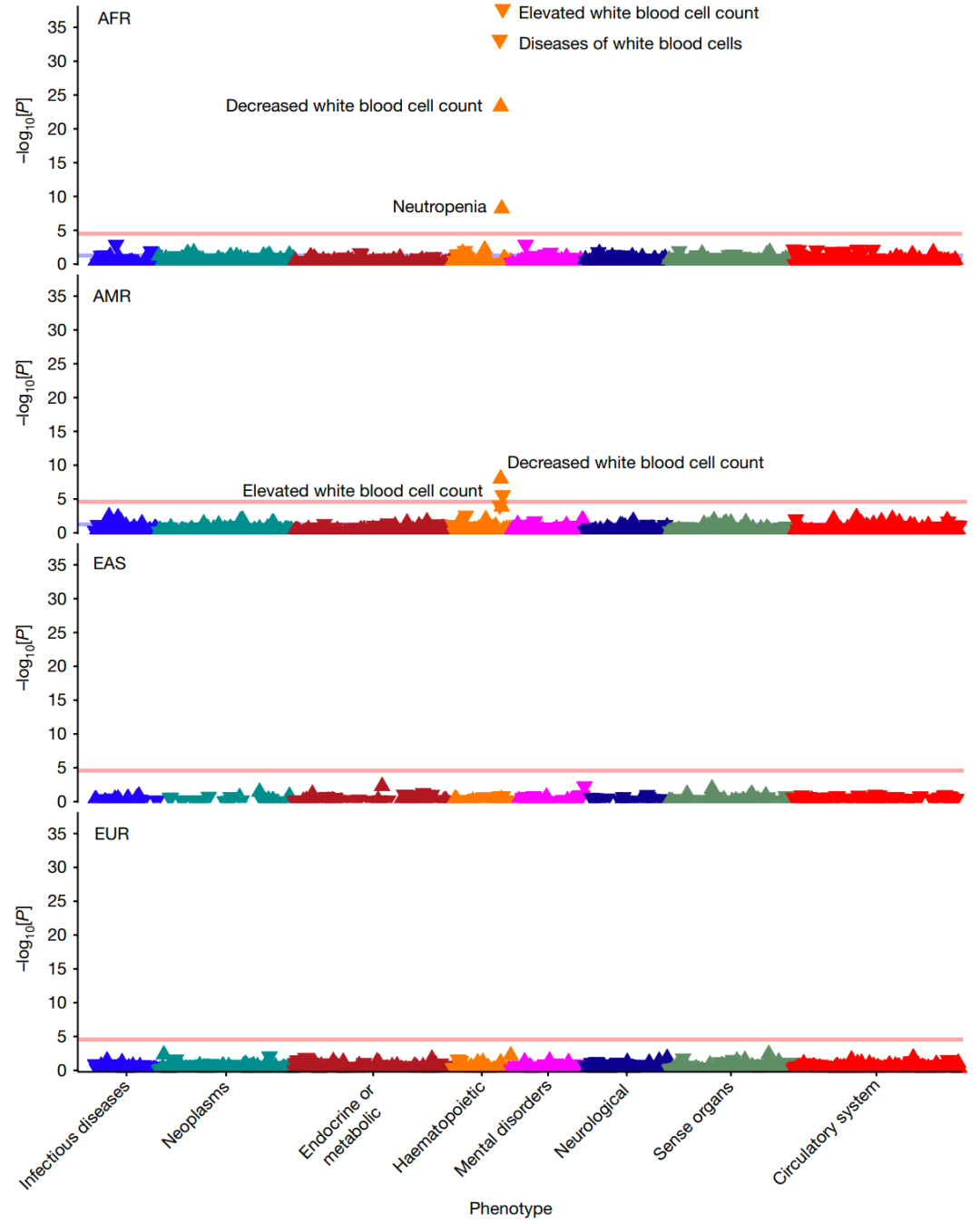
图4. 达菲血型位点的全表型关联分析
结 语
该文章描述了All of Us研究计划的方法,其以前所未有的规模产生多样化的临床级基因组数据,共发布了大约245000个基因组序列的数据。研究团队进行了一系列数据整合和质量控制程序,并对数据集的特征进行了分析,包括遗传祖先和亲缘关系,并通过重现包括LDL-C和117种其他疾病在内的已建立的基因型-表型关联来验证该数据集。All of Us进行的临床级测序不仅可以进行研究,还可以通过临床相关的遗传结果和与健康相关的特征为选择接收这些信息的参与者提供有价值的回馈。因此,在未来,这一数据资源将大大促进基因组学领域的发展。
论文原文:
The All of Us Research Program Genomics Investigators. Genomic data in the All of Us Research Program. Nature (2024). https://doi.org/10.1038/s41586-023-06957-x.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









#基因组数据# #全基因组序列# #编码变异#
73