Nature Genetics:DeepLoop:利用低深度Hi-C数据绘制人类基因组等位基因特异性染色质互作图谱
2022-08-09 测序中国 测序中国
研究团队开发了DeepLoop,执行严格的偏差校正,通过基于深度学习的信号增强,从低深度Hi-C数据中实现稳健的染色质相互作用映射。
Hi-C技术能够可靠地识别三维基因组特征,如区隔和拓扑相关域(TADs),改变了人们对哺乳动物基因组的认识。但是当分辨率在千基尺度(kilobase)时,复杂的结构和数据分布使Hi-C接触热图变得嘈杂。迄今为止,染色质环的全基因组图谱,特别是TADs中的增强子启动子相互作用,仍然是Hi-C分析的一个主要挑战。因此,科学家们开发了相关技术,比如通过对端标签测序(ChIA-PET)分析染色质相互作用,启动子捕获Hi-C(pcHi-C), HiChIP/邻近连接辅助芯片- seq(place - seq)等,以在靶标位点获得更好的信噪比。
近日,来自美国克利夫兰凯斯西储大学医学院的研究团队开发了基于深度学习的信号增强策略DeepLoop,用于从低深度Hi-C数据进行稳健的染色质相互作用映射。研究表明,DeepLoop能够绘制人类基因组中等位基因特异性染色质相互作用的遗传和表观遗传决定因素,并在低深度Hi-C数据调用循环中实现超高的鲁棒性,支持单细胞Hi-C分析。研究团队将研究结果发表Nature Genetics上,文章题为“DeepLoop robustly maps chromatin interactions from sparse allele-resolved or single-cell Hi-C data at kilobase resolution”。

研究团队利用自动编码器对高深度Hi-C热图进行去噪(图1),在人胎儿大脑皮层中选取3个重复的Hi-C数据进行模型训练,每个重复中有1.4 ~ 1.5亿个顺式触点(<2 Mb)。将HiCorr(Hi-C bias-correction pipeline)应用于每个重复,并在片段分辨率(~5 kb)下提取~18000个子矩阵作为训练集。正如先前报道的那样,HiCorr生成的距离校正热图比ICE/KR9更清晰,但噪声像素仍然存在。由于在生物重复之间,真实的循环像素比噪声像素更具有可重复性,研究团队通过只保留可重复性循环像素来建立“训练目标”热图。
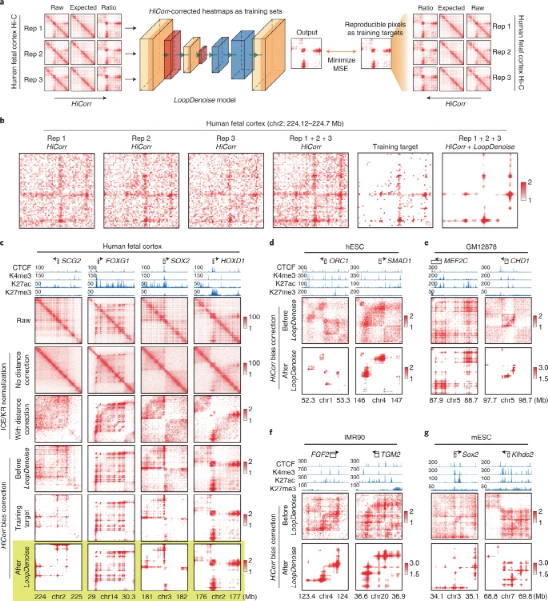
图1. HiCorr和LoopDenoise从嘈杂的Hi-C数据集显示染色质环。
随后,研究团队开发了一种分析低深度Hi-C数据的方法,训练了一系列U-Net38 LoopEnhance模型,其中有大约10 - 2.5亿个中端顺式接触。研究团队使用高深度数据的loop去噪输出作为训练目标(图2)。虽然当读取深度小于50 Mb时环路信号很难识别,但从低深度Hi-C数据得到的增强热图几乎是相同的。当将增强的热图与完整数据(~ 3.8亿个中端顺式接触)进行比较时,发现当读取深度降低到1亿个时,性能没有下降(像素级再现性>70%),即使测序深度降低到1250万,像素级的重复性仍保持在>50%。研究团队还用来自H9 hESCs的Hi-C数据训练了新的DeepLoop模型(LoopDenoise和LoopEnhance),并证实训练集的选择不影响结果。由于DeepLoop热图中的像素强度代表了Hi-C信号的富集,研究团队可以直接将顶部循环像素作为相互作用。需要注意的是,DeepLoop不输出一个明确的离散“循环”列表,将“循环像素”转换为“循环”需要新的算法和参数,这将不可避免地引入新的偏差。因此,研究团队保留了DeepLoop作为“what-you-see-is-what-you-get”方法。

图2. LoopEnhance实现了从低深度Hi-C数据进行灵敏、鲁棒的环路调用。
为了进一步探索跨平台一致性,研究团队比较了HindIII、DpnII和micro-C9制备的H1 hESCs中已发表的超深Hi-C数据。无论读取深度如何,来自HindIII和DpnII Hi-C的DeepLoop热图在像素级别上更加相似。当消解分辨率提高时(从Hi-C到micro-C),KR-ratio热图变得更清晰,更类似于DeepLoop输出。当以micro-C KR-ratio热图为参考比较其他信号增强方法时,DeepLoop显示出最高的相关系数。DeepLoop增强的低深度(5000万)Hi-C数据性能优于所有其他分析框架,甚至优于KR处理的全深度数据。
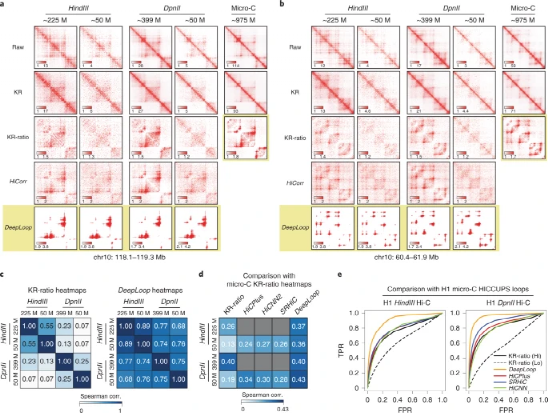
图3.无论读取深度和消解分辨率如何,DeepLoop都输出收敛的Hi-C环路剖面。
最后,研究团队分析了杂合SNP对染色质环的影响。在排除与印迹、X -失活和SVs相关的AS循环像素后,利用简单的双重截断在1959个位点调用数千个AS循环像素(图4),包含91304个杂合SNP。结果显示,CTCFL和CTCF是在loop阳性等位基因中富集最多的两个基序,证明DeepLoop解析loop遗传的可行性。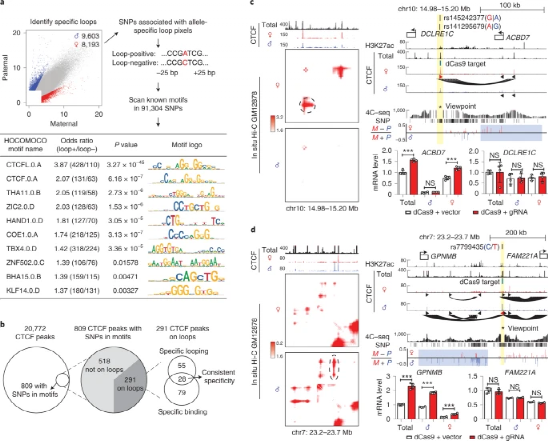
图4.等位基因DeepLoop图谱精确定位影响染色质环的常见SNP。
综上所述,研究团队开发了DeepLoop,执行严格的偏差校正,通过基于深度学习的信号增强,从低深度Hi-C数据中实现稳健的染色质相互作用映射。DeepLoop可以实现环路分辨率、单细胞Hi-C分析,还能实现不同Hi-C协议和micro-C之间的跨平台一致性。DeepLoop可以精确定位杂合子单核苷酸多态性和大型结构变异,扩展了Hi-C的使用,为三维基因组遗传学研究提供了千碱基分辨率的分析方法。
参考文献:
Zhang, S., Plummer, D., Lu, L. et al. DeepLoop robustly maps chromatin interactions from sparse allele-resolved or single-cell Hi-C data at kilobase resolution. Nat Genet 54, 1013–1025 (2022). https://doi.org/10.1038/s41588-022-01116-w
https://www.nature.com/articles/s41588-022-01116-w
·END ·
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言













#Genetics#
84
#Gene#
65
#Nat#
54
#Genet#
54
#NET#
68
#等位基因特异性#
51
#特异性#
96
#互作#
64
#等位基因#
54
#DeepL#
90