bioRxiv:新冠病毒检测和研究的三种测序方法对比
2020-03-26 MGI华大制造 MGI华大制造
随着新冠病毒的全球肆虐,如何在针对疑似样本的大规模检测中,既能提高检测灵敏度,又能获得病毒全基因组序列以用于后续变异监测和研究。
随着新冠病毒的全球肆虐,如何在针对疑似样本的大规模检测中,既能提高检测灵敏度,又能获得病毒全基因组序列以用于后续变异监测和研究,中国科学家于2020年3月16日预印在bioRxiv的一篇文章《Multiple approaches formassively parallel sequencing of HCoV-19 genomes directly from clinical samples》给出了答案。
文章分别采用宏基因组测序(Meta)、探针捕获测序(Capture)和多重PCR捕获测序(Amplicon)等三种方法对8例梯度稀释的病毒培养物(Ct值17.3-39.9)和8例新冠病毒临床样本(Ct值18-32,包括咽拭子,喉拭子,鼻拭子,肛拭子和唾液样本)进行了检测对比。首先采用宏基因组方法对临床样本进行了超高通量测序,每个样本产出>300Gb数据,其中病毒reads占总reads比例仅0.002%-5.513%。
样本数据量要求
在同时满足全基因组95%覆盖度以及10X测序深度的情况下:在病毒梯度稀释样本中(102-106copies/ml)进行探针捕获测序需要5.3Mb-14Gb数据,进行多重PCR捕获测序需要33Mb-973Mb数据;在临床样本中,进行探针捕获测序需要12Mb-25Gb数据,进行多重PCR捕获测序需要32Mb-1.8Gb数据,而宏基因组需要129Mb-334Gb数据。
表1一定深度下三种方法所需要的数据量

满足高要求下游分析的情况下(捕获≥20X,多重≥100X,宏基因组≥10X,覆盖度≥95%),在病毒梯度稀释样本中(102-106copies/ml),捕获需要8Mb-24Gb数据,多重需要185Mb-2.7Gb数据;在临床样本中,捕获需要23Mb-25Gb数据,多重需要261Mb- 1.8Gb数据,而宏基因组需要129Mb-334Gb数据。
表2满足下游深度分析三种方法所需要的数据量
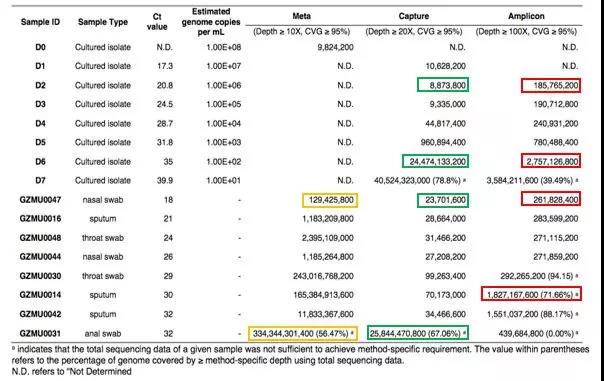
通过对比可知,多重PCR捕获测序方法在不同病毒载量样本中所需要的数据量差异更小,在低病毒载量要求的数据量更少。
病毒载量检测灵敏度
采用华大智造SARS-CoV-2 full length genome panel试剂盒,对梯度稀释的病毒培养物(Ct值17.3-39.9)和临床样本(Ct值18-32,包括咽拭子,喉拭子,鼻拭子,肛拭子和唾液样本)进行检测对比,结果表明:对于高病毒载量(≥105copies/ml),探针捕获测序和多重PCR捕获测序都有很好的富集效果(相对于宏基因组方法分别获得了5596倍和5710倍富集);而在低病毒载量的样本中(≤104copies/ml,Ct值≥28.7)多重PCR捕获测序的富集效果是探针捕获测序的10-1000倍。这一结果表示,在低病毒载量的情况下,多重PCR捕获测序具有更好的检测灵敏度。
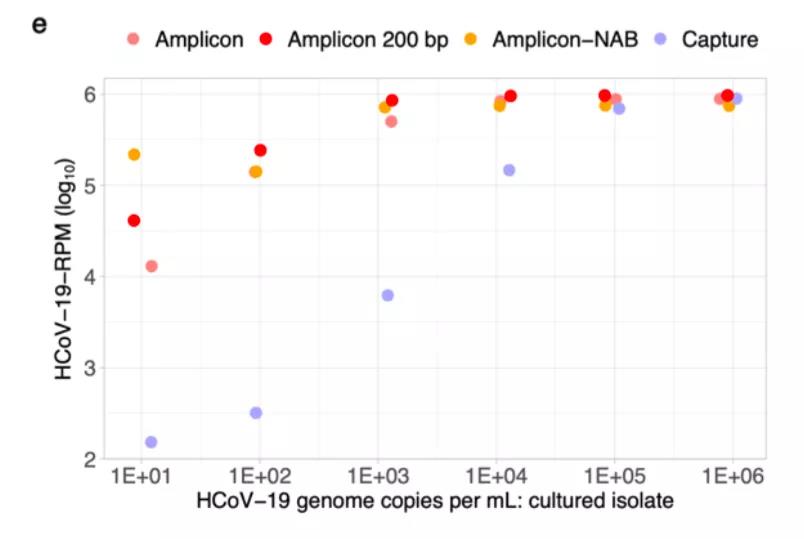 #RPM-每M数据中病毒的数据的比例
#RPM-每M数据中病毒的数据的比例
图1三种方法对病毒富集的效果
突变检测准确性
在临床样本中,基于多重PCR捕获测序和宏基因组测序方法都检测到19个突变位点,而探针捕获测序只检测到18个位点,漏检一个点,并且该位点对应的样本病毒载量低(Ct 值30)。这一结果表明多重PCR捕获测序方法相对更为准确,特别是在极端样本情况下。
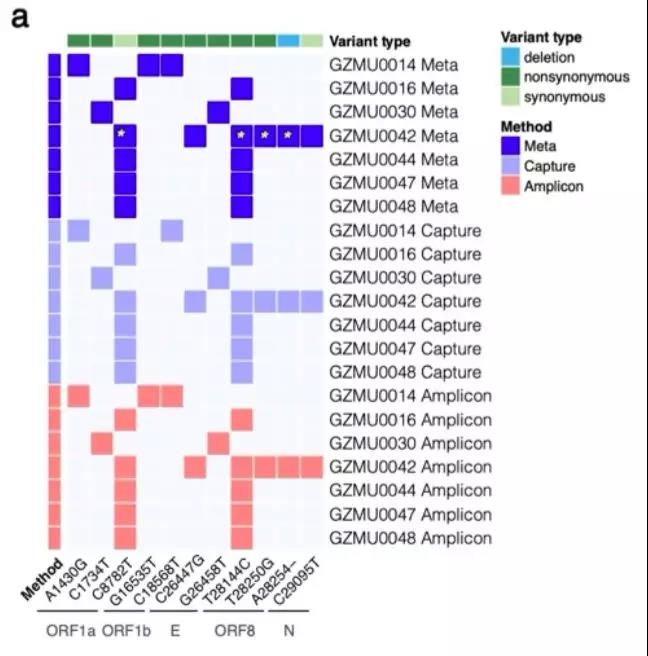
图2 三种方法突变检测结果
结论
综上,基于高通量测序的三种方法选择指导:
1.如果研究者想要研究除目标微生物外其他的病原微生物,并且病毒载量≥ 105copies/ml或Ct值≤ 24.5,推荐宏基因组测序;2.如果研究者只想研究目标微生物,比较有挑战的样本譬如病毒载量<105 copies/ml或Ct值>24.5,推荐探针捕获测序(关注次要碱基突变和碱基频率)和多重PCR测序(关注主要碱基突变);3.如果研究者只想研究目标微生物,并重点关注主要碱基突变,样本极具挑战性,病毒载量低至102copies/ml或Ct值高至35,推荐多重PCR测序。
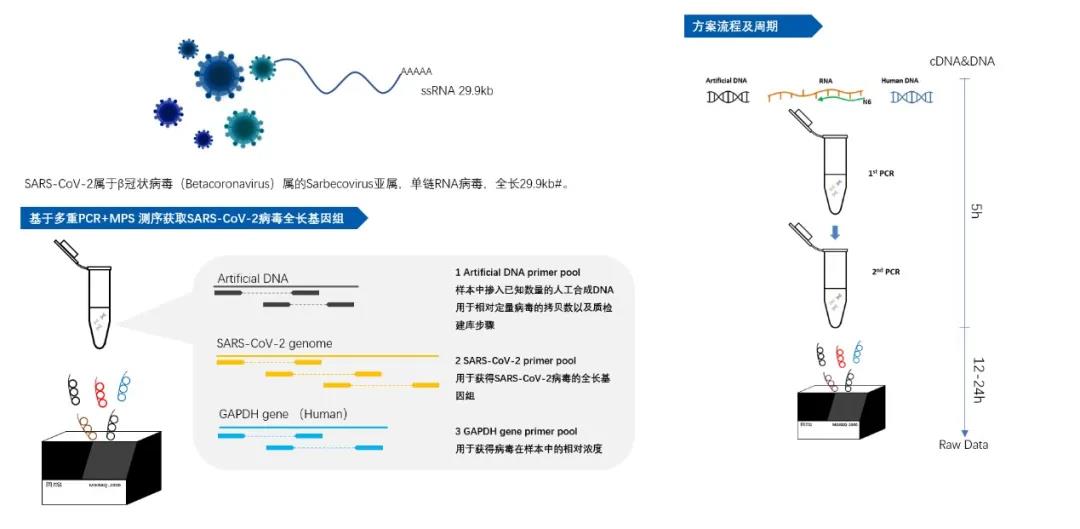
目前,针对以上三种方法,华大智造DNBSEQ平台均已推出配套整体解决方案。特别地,在该文中采用的SARS-CoV-2 full length genome panel试剂盒基于华大智造ATOPlex多重定制化服务平台打造。该试剂盒除了应用于全基因组组装、病毒变异检测、进化研究外,还能够应用于极端新冠样本的检测,最低仅需1M Reads就可获取病毒全基因组信息,其表现出比常规RT-PCR更高的灵敏度,能够满足RT-PCR假阴性样本的二次检测、复核确诊和大规模疑似样本检测的需要。
关于SARS-CoV-2 full length genome panel试剂盒
基于锁定引物的2步PCR,特异性强,中间不需要额外的消化步骤,操作简单;能够在一管中实现病毒全基因的扩增,不需要分管操作;加入已知拷贝数人工合成外参DNA,并同时进行病毒基因和外参DNA的检测,用于病毒的载量确定;加入看家基因检测,可以用于病毒载量相对于细胞含量的确定,有助于病毒细胞病理学研究;双唯一barcode结合DNBSEQ测序,最大程度降低barcode串扰,降低平行测序过程中强阳性样本对弱阳性或阴性样本的污染。
关于ATOPlex多重PCR定制化服务平台
ATOPlex多重PCR定制化服务平台是华大智造自主研发的以超高重PCR技术为核心的产品定制平台。该平台涵盖了DNA、RNA和DNA甲基化方向,可以满足科研、医药、司法、农业、DTC等相关领域的个性化基因检测产品定制。ATOPlex多重PCR定制产品平台提供产品设计、建库试剂,此外,还可以提供该产品基于MGISP-100或者MGISP-960的自动化建库解决方案。
原始出处:
Minfeng Xiao, Xiaoqing Liu, Jingkai Ji, et al. Multiple approaches for massively parallel sequencing of HCoV-19 (SARS-CoV-2) genomes directly from clinical samples. bioRxiv 2020.03.16.993584; doi: https://doi.org/10.1101/2020.03.16.993584.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言







#Bio#
65
#新冠病毒检测#
103
#病毒检测#
66
新冠肺炎,疫情何时才能消失
72