PNAS:丙肝病毒通过劫持肝脏microRNA以幸存
2012-01-20 MedSci MedSci原创
病毒性疾病仍然是医学科学的最大挑战之一。由于与人类数千年的协同进化,它们利用人类宿主生物学生存繁荣的能力使他们很难成为医学治疗的靶标。 北卡罗来纳大学(UNC)教堂山分校的科学家们与科罗拉多大学的同事们一起研究,首次证明一个人类肝细胞中调节基因表达的小RNA分子如何被丙型肝炎病毒劫持以确保此病毒的幸存--帮助医学家们理解为什么一个新的抗病毒药物出现有效的抗病毒作用。 Micro
病毒性疾病仍然是医学科学的最大挑战之一。由于与人类数千年的协同进化,它们利用人类宿主生物学生存繁荣的能力使他们很难成为医学治疗的靶标。
北卡罗来纳大学(UNC)教堂山分校的科学家们与科罗拉多大学的同事们一起研究,首次证明一个人类肝细胞中调节基因表达的小RNA分子如何被丙型肝炎病毒劫持以确保此病毒的幸存--帮助医学家们理解为什么一个新的抗病毒药物出现有效的抗病毒作用。
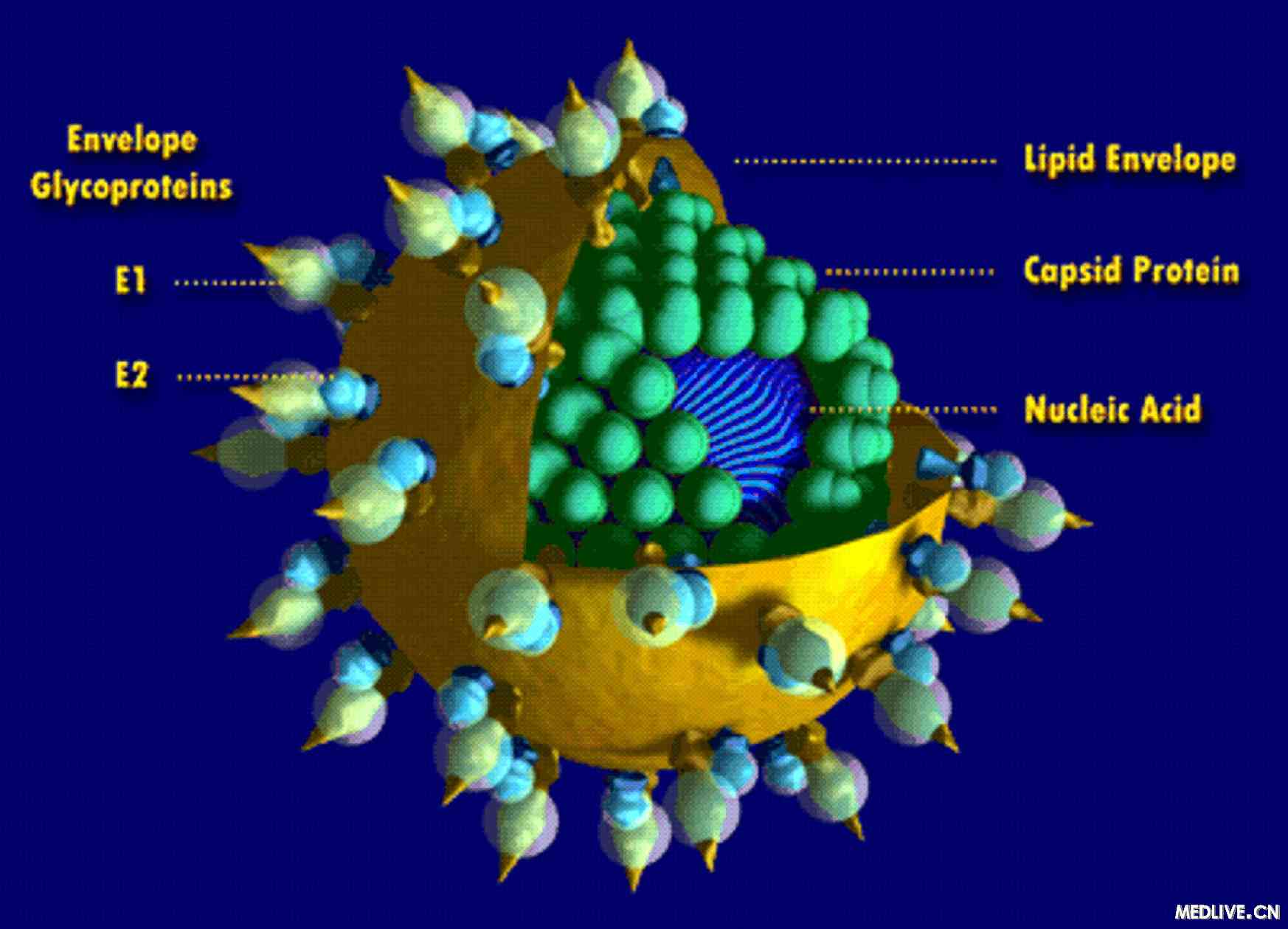
MicroRNAs涉及调节细胞内基因表达,通常通过阻断关键蛋白生产或通过使细胞生长和分化时编码细胞蛋白的信使RNA不稳定的方式。正常地,它们通过下调基因表达发挥作用。研究小组发现,肝细胞内名为miR-122的microRNA与病毒RNA的结合可导致病毒稳定,促进病毒基因组在肝脏的有效复制,支持病毒的生命周期。
"丙型肝炎病毒与miR-122一起做两件非常有趣的事",Stanley M. Lemon博士说,他是医学微生物学与免疫学教授,北卡罗来纳大学综合癌症中心和移植免疫学中心的成员。
"首先,它进化了与关键调节子的一种独特的关系,因为miR-122代表了存在于肝脏中约一半的microRNA。其次,该病毒已篡夺了通常下调基因表达来上调其RNA稳定性的过程,表达其生命周期所需要的病毒蛋白。这是病毒如何为它们自己邪恶目的而颠覆细胞正常有益功能的一个典型例子。"
由Lemon博士和他的同事在2005年所做的工作有助于示范miR-122是丙型肝炎病毒复制自己所必需的,但机制尚不清楚。现在,UNC的研究小组已经指出它是如何工作的,这有助于解释一个新的抗病毒药物如何瞄准病毒。称为antagomer的药物,在肝中结合并分离miR-122,然后使病毒基因组不稳定,加速其在肝脏中降解。最新的研究结果发表在本周的《PNAS》( Proceedings of the National Academy of Sciences)上。
丙型肝炎是一个持续的公共健康问题,这个病难以测定,因为症状在感染后几个月到几年后出现。疾病控制与预防中心(Centers for Disease Control and Prevention)估计,美国多达400万人可能长期感染丙型肝炎病毒,而且大多数人不知道他们感染此病毒。超过三分之一的长期携带者可能发展慢性肝病或肝癌,这是一种致命性癌症,它因为这种病毒的传播而正变得日益普遍。(生物谷bioon.com)
MicroRNA-122 antagonism against hepatitis C virus genotypes 1-6 and reduced efficacy by host RNA insertion or mutations in the HCV 5′ UTR
Yi-Ping Li, Judith M. Gottwein, Troels K. Scheel, Tanja B. Jensen, and Jens Bukh
Abstract MicroRNA-122 (miR-122) is believed to stimulate hepatitis C virus (HCV) replication through interaction with two adjacent sites downstream of stem loop I (SLI) within the HCV 5′ untranslated region (5′ UTR). Recently, it was demonstrated that locked nucleic acid SPC3649-induced miR-122 antagonism suppressed HCV genotype 1a and 1b infection in vivo. However, virus-producing culture systems with 5′ UTR of different HCV genotypes have not been available for testing 5′ UTR-based treatment approaches. Using JFH1-based Core-NS2 genotype recombinants, we developed 5′ UTR-NS2 recombinants of HCV genotypes 1a, 1b, 2a, 2b, 3a, 4a, 5a, and 6a with efficient growth in Huh7.5 cells. Deletion mutagenesis studies demonstrated that the 5′ UTR SLI was essential for genotypes 1-6 infection. However, lack of SLI could be compensated for by insertion of other structured HCV or host RNA sequences, including U3 small nucleolar RNA. We demonstrated that SPC3649-induced miR-122 antagonism had a potent antiviral effect against HCV genotypes 1-6 5′ UTR-NS2 viruses. Strikingly, HCV recombinant virus with substitution of SLI and miR-122 binding site 1 (S1) by the U3 RNA sequence was not affected by miR-122 antagonism; this was attributed to the lack of an intact S1 by reverse genetics studies. Therefore, we engineered the corresponding U3 RNA sequences into S1 and demonstrated that HCV recombinants with wild-type SLI and single or combined mutations at four of eight nucleotides of S1 were viable in Huh7.5 cells. These mutations reduced the efficacy of SPC3649 treatment, indicating that escape variants to miR-122 antagonism-based HCV therapy could potentially occur.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言










#Micro#
68
#CRO#
63
#PNAS#
69
#microRNA#
71
#丙肝病毒#
88