Nature:从随机搜索到精准激活:翻译起始的新机制
2024-12-15 生物探索 生物探索 发表于陕西省
本文介绍解析 mRNA 帽端识别机制研究,用 smFRET 技术揭示 eIF4F 动态行为,提出新模型,填补空白,且为肿瘤、病毒感染等疾病治疗提供新思路。
引言
基因表达是维持生命活动的核心机制,而翻译起始则是其中的重要环节。翻译起始过程的效率和准确性直接影响蛋白质的合成,这对细胞的正常功能至关重要。在真核生物中,大多数mRNA分子需要依赖位于5’端的7-甲基鸟苷帽(7-methylguanosine cap,简称mRNA帽端)才能启动翻译。然而,这一关键步骤的具体分子机制长期以来一直是研究中的热点和难点。
过往的研究表明,一个名为eIF4F的三聚体复合物在识别mRNA帽端并激活翻译方面扮演着重要角色。eIF4F由三个亚基组成:负责直接结合帽端的eIF4E、具有解旋酶功能的eIF4A以及作为支架蛋白的eIF4G。这些亚基共同协作,帮助将mRNA招募到核糖体预起始复合物中,从而开启蛋白质的合成。然而,尽管研究人员已经对这些分子进行了几十年的研究,eIF4F如何高效而准确地识别帽端,以及其如何完成mRNA激活这一关键步骤,仍然存在许多未解之谜。
12月11日Nature的研究报道“The mechanism of mRNA cap recognition”针对这一领域的空白,开发了一种单分子荧光共振能量转移(smFRET)技术,通过实时观察单个分子的行为,详细解析了eIF4F复合物在mRNA上的动态行为。研究发现,eIF4F不仅能够随机探索mRNA的不同位置,还能通过其内部亚基间的协作,在识别帽端时表现出独特的“选择性”和“活化”模式。此外,该研究还揭示了eIF4A在去除非合成性结合的eIF4F复合物中的关键作用,以及eIF4E与帽端结合时如何稳定整个复合物。这一研究不仅填补了翻译起始机制的空白,也为开发针对翻译调控的潜在疾病治疗手段提供了全新的思路。

从生命密码到蛋白质工厂:mRNA帽端的神秘角色
DNA蕴藏着生命活动的密码,但要将这些密码转化为功能多样的蛋白质,翻译这一过程至关重要。而在这个过程中,mRNA(信使RNA)扮演着不可或缺的桥梁角色。它不仅将DNA的遗传信息传递到蛋白质合成的“工厂”——核糖体,还需要精确的调控来确保基因表达的效率与准确性。然而,这一过程的核心玩家之一——mRNA的7-甲基鸟苷帽端(7-methylguanosine cap,简称帽端)却隐藏着复杂而神秘的作用机制。
mRNA帽端是每条真核生物mRNA在其5’端所携带的一个化学结构,它不仅为mRNA提供稳定性,防止其被核酸酶降解,还为核糖体的招募提供了信号。据统计,超过95%的真核细胞mRNA都依赖帽端参与的翻译起始过程来实现蛋白质的合成。而帽端的重要性不仅在于其物理结构,更在于它所参与的精确分子机制,这直接影响了细胞内的基因表达调控。研究显示,帽端在翻译起始中的功能犹如一位“通行证检查员”,其存在决定了一条mRNA是否能够顺利进入核糖体进行翻译。
尽管帽端的基本功能早已为人所知,但其具体的作用机制却始终未被完全揭示。例如,负责识别帽端的关键分子——真核翻译起始因子4F复合物(eIF4F)的运作方式,长期以来都被认为是一个多步复杂的过程。然而,这种识别是如何实现的?分子之间如何协作完成这场精准的“帽端搜索”?这些问题悬而未决。
正因如此,关于mRNA帽端的研究成为分子生物学的一个重要领域。解开这个机制之谜,不仅有助于我们深入理解基因表达的核心过程,还可能为治疗与翻译调控相关的疾病(如癌症、病毒感染)提供新的靶点。
“翻译守门人”:eIF4F复合物的复杂协作
eIF4F,这个由三个亚基组成的分子机器通过协同合作,确保mRNA成功加载到核糖体,从而开启蛋白质的翻译过程。
eIF4F复合物的三个亚基分别是eIF4E、eIF4A和eIF4G。它们各司其职,又密切协作。eIF4E是一个小型帽端结合蛋白,能够特异性地识别并与mRNA的7-甲基鸟苷帽端结合,是整个复合物的“起点”。eIF4G则是复合物的核心支架蛋白,不仅连接eIF4E和eIF4A,还可能与其他翻译因子相互作用,将mRNA与核糖体的43S预起始复合物连接起来。eIF4A则是一种DEAD-box RNA解旋酶,通过水解ATP提供能量,解开帽端附近的RNA二级结构,从而让核糖体更顺畅地进入。
然而,这三个亚基的协作并非机械式的简单叠加,而是通过精细的动态调控实现的。研究发现,eIF4E与帽端的结合可以通过eIF4G进一步稳定,同时引导eIF4A在正确的区域展开解旋活动。而这种协作不仅提高了翻译效率,也赋予了翻译起始过程高度的特异性。尽管eIF4F的工作模型已经被研究人员广泛接受,但对于每个亚基在翻译起始过程中的具体功能和分子作用机制,仍有许多未解之谜。
突破技术限制:单分子荧光技术的应用
研究分子层面上的翻译起始机制是一项复杂的挑战。传统的生化实验方法通常依赖于大规模样本的平均表现,这使得揭示分子之间的动态相互作用变得困难。而在探索mRNA帽端(cap)识别机制和翻译起始因子4F复合物(eIF4F)行为时,这一技术局限性显得尤为突出。为了解决这一难题,研究人员引入了单分子荧光共振能量转移(smFRET)技术,将分子层面的精细动态变得可视化。
smFRET是一种基于荧光信号的技术,通过标记分子上的特定位点,研究人员能够实时观察单个分子在其环境中的运动和相互作用。在该研究中,研究人员将荧光标记分别放置在mRNA分子的特定位置(靠近或远离帽端),以及eIF4F复合物的关键亚基eIF4G上。通过这种方法,他们能够精确测量eIF4F复合物在mRNA上的结合和解离动态,甚至细致到不同位置的结合速率和持续时间。
相比于传统的聚合实验,smFRET的优势在于其“单分子分辨率”。通过超过60,000个分子的观察和超过80种实验条件的测试,揭示了eIF4F复合物的动态行为。
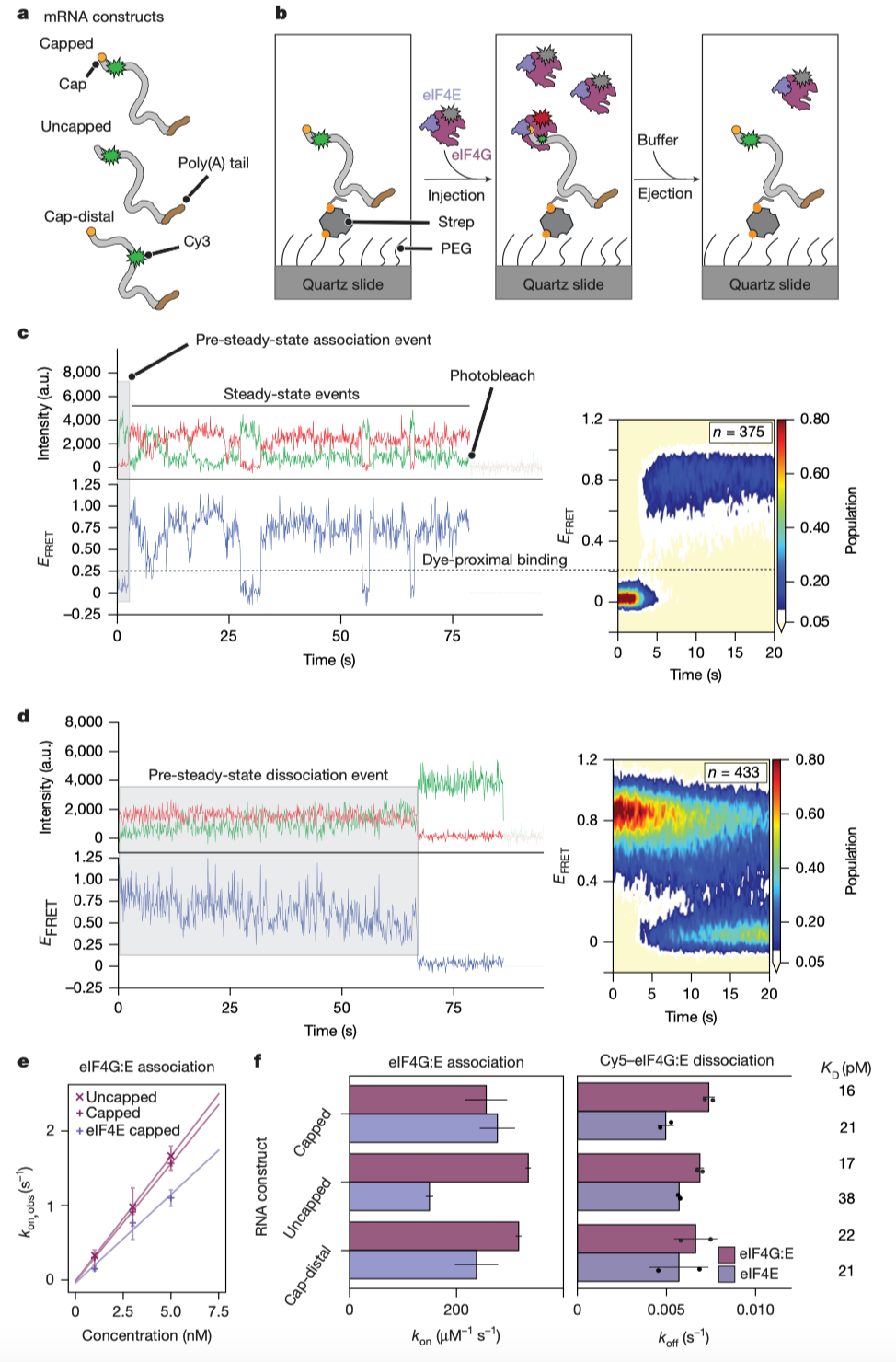
eIF4G及其与eIF4E结合形成的复合物(eIF4G:E)如何与mRNA结合的实验设计与主要结果(Credit: Nature)
图a. 研究使用了完整长度的rpl41a mRNA,并在不同位置(靠近帽端或远离帽端)进行了Cy3荧光标记,生成三种实验构建体:帽端附近标记的带帽mRNA、帽端附近标记的不带帽mRNA,以及帽端远离位置标记的带帽mRNA。这些标记构建体为分析eIF4G的结合位置和行为提供了实验基础。
图b. 通过停流注射与排出实验(stopped flow injection and ejection experiments),研究人员分别测量了eIF4G或eIF4G:E与mRNA结合的动力学参数,包括结合速率常数(k on)和解离速率常数(k off )。注射实验用于观察分子结合的初始阶段,排出实验则通过移除多余未结合蛋白来研究结合的稳定性。
图c, d. 展示了Cy5标记的eIF4G:E与mRNA结合和解离的典型轨迹:图c中的注射实验显示了eIF4G:E的结合行为,可分为两个阶段:初始的非稳态结合阶段(pre-steady-state)和后续的稳态绑定与解离过程,直到荧光团光漂白。图d中的排出实验则仅能反映非稳态绑定行为,因为流室中已移除未结合的蛋白,因此无稳态数据。
图e. 揭示了eIF4G:E的结合速率与蛋白浓度呈线性关系,但结合速率更多依赖于eIF4E的存在,而不是mRNA的帽端结构。这说明eIF4E对eIF4G:E的绑定效率有明显促进作用。
图f. 对注射和排出实验中的结合和解离速率进行了量化。结果表明,无论帽端是否存在或结合位置如何,eIF4G对mRNA的结合都是均匀且稳定的。这表明eIF4G在结合行为上缺乏对帽端的特异性。
“随机搜索”到精准识别:eIF4F如何找到帽端?
通过单分子荧光共振能量转移(smFRET)技术,研究人员首次揭示了eIF4F在mRNA上的动态行为,展现了从“随机搜索”到“精准识别”的奥妙过程。
研究发现,eIF4F复合物的识别并非一开始就锁定帽端,而是通过eIF4G在mRNA上的随机结合实现的。smFRET实验显示,eIF4G在不同位置上的结合和解离是随机的,其结合速率和持续时间并不受帽端存在的影响。这表明,在初始阶段,eIF4F复合物并未表现出对帽端的特异性。这种“随机搜索”策略让eIF4F能够快速扫描整条mRNA,而不浪费时间在特定位置停留。
当帽端存在时,情况发生了转变。eIF4E通过其帽端结合能力触发了一系列协同机制,使得eIF4F复合物从不稳定的结合状态过渡到稳定的结合状态。研究显示,这种稳定化过程依赖于eIF4G与eIF4E之间的协作:eIF4E首先识别帽端,随后通过与eIF4G的相互作用进一步加强了整个复合物的帽端结合能力。此外,帽端识别还通过抑制eIF4A的“清道夫”功能,确保已经结合帽端的复合物不会被移除。
令人惊讶的是,即使在帽端附近,eIF4F仍需要一定的动力学调整。研究显示,帽端的识别并非完全由化学结合决定,还涉及复合物内部亚基间的构象变化。这种动态机制表明,eIF4F复合物不仅是一个简单的分子“锁”,更是一个动态响应的“调控中心”。
通过这些发现,研究人员为帽端识别过程提出了一种新模型:eIF4F复合物通过随机探索确保效率,通过帽端结合触发稳定性,从而完成了从随机搜索到精准识别的飞跃。
eIF4A的“清道夫”角色:驱动帽端识别的隐藏力量
在翻译起始的过程中,eIF4A常常被认为是一个辅助角色,其主要功能是作为RNA解旋酶清除帽端附近的二级结构。然而,该研究揭示了eIF4A的另一种关键功能——“清道夫”角色,它通过去除非合成性结合的复合物,优化了eIF4F在mRNA帽端的精准识别能力。
研究表明,eIF4G可以与mRNA的多个位置发生非特异性结合,包括远离帽端的区域。这些非合成性结合尽管有助于初期的随机搜索,但也可能阻碍eIF4F复合物精准识别帽端的效率。此时,eIF4A的“清道夫”作用显现出来。通过单分子荧光共振能量转移(smFRET)技术,研究人员发现,eIF4A在ATP水解的驱动下,能够有效地从mRNA上剥离非合成性结合的eIF4G。这一过程确保了eIF4F复合物不会长期停留在错误的位置,为其探索帽端创造了更大的动态空间。
令人惊讶的是,这种“清道夫”功能具有高度选择性。当eIF4F复合物最终接近帽端时,帽端与eIF4E的结合会通过eIF4G产生一种全新的构象变化,这种变化抑制了eIF4A的清除活性,保护已经结合帽端的复合物免受进一步移除。这种巧妙的机制不仅提高了帽端识别的效率,还减少了识别过程中的资源浪费。
更进一步的研究还发现,eIF4A的活性依赖于与eIF4G的直接相互作用,而非简单的RNA解旋功能。通过调控ATP的结合与水解,eIF4A能够在不同的mRNA区域间动态跳跃,同时保持对帽端附近复合物的稳定性。这种“择优而去”的行为模式,恰如一名高效的清道夫,帮助eIF4F复合物精准定位其真正的目标。
这项发现为eIF4A在翻译起始中的角色赋予了新的意义。它不仅是一个RNA解旋酶,更是优化帽端识别的关键驱动力。
翻译起始的“激活态”:一个新模型的提出
该研究通过单分子荧光技术,提出了一个全新的分子模型,重新定义了mRNA的激活过程,为我们理解翻译调控打开了新的视角。
研究发现,mRNA激活并非简单的帽端识别,而是一个动态的、多步骤的过程。在这个过程中,eIF4F复合物(由eIF4E、eIF4G和eIF4A组成)发挥了核心作用。首先,eIF4G以随机搜索的方式在mRNA上结合多个位置,包括帽端附近和远离帽端的区域。随后,通过eIF4A的“清道夫”功能,非合成性结合的复合物被移除,确保eIF4F能够继续寻找帽端。
当eIF4F复合物到达帽端时,eIF4E与帽端的结合触发了复合物内部的一系列全新构象变化。这种变化不仅进一步稳定了帽端的结合,还通过eIF4G向eIF4A发出“停止清除”的信号,从而保护eIF4F复合物在帽端的结合。这一状态被研究人员称为“激活态”,它标志着mRNA已准备好与43S核糖体预起始复合物结合,并完成翻译起始的关键步骤。
与传统的“帽端即激活”模型相比,这一新模型强调了动态调控的作用。eIF4F复合物的随机搜索、帽端结合触发的构象变化以及eIF4A的精准调控共同构成了这一精妙的分子机器。这种动态性确保了翻译起始过程的高效性与特异性,同时赋予了细胞根据环境变化调控基因表达的灵活性。
这一模型的提出,改变了我们对翻译调控的认识:翻译起始不仅依赖于分子之间的化学结合,还受到动态相互作用和协同性的高度调控。
从基础科学到临床应用:未来的无限可能
mRNA帽端识别机制的研究不仅解开了翻译起始这一核心生物学过程的分子奥秘,还为临床医学的应用带来了令人振奋的可能性。
在肿瘤研究领域,eIF4F复合物的异常活性被认为与癌细胞的快速增殖密切相关。许多肿瘤通过过表达eIF4E或改变eIF4G的调控机制来增强翻译效率,从而促进癌症相关基因的表达。基于这一研究揭示的分子机制,靶向eIF4F复合物的药物设计将更加精准。例如,通过抑制eIF4E与帽端的结合或干扰eIF4A的“清道夫”功能,可以有效阻断癌细胞对翻译起始过程的依赖,从而抑制肿瘤的生长。
此外,在病毒感染的背景下,这一研究的潜在应用同样令人期待。许多病毒依赖宿主细胞的翻译起始机制来合成其蛋白质。例如,病毒可以通过绑架eIF4F复合物来优先翻译病毒基因组。通过调控eIF4F的动态行为或干扰帽端识别机制,研究人员有望开发针对病毒复制的新型抗病毒疗法。这种方法不仅具有高效性,还能最大限度减少对宿主细胞正常翻译的影响。
更广泛地,这一研究为理解翻译调控在其他疾病中的作用提供了新思路。无论是神经退行性疾病还是代谢紊乱,翻译起始因子的异常活性都可能在疾病发生中扮演重要角色。未来,通过深入研究这一机制并开发相应的干预手段,有望为个性化治疗方案奠定基础。
参考文献
Gentry, R.C., Ide, N.A., Comunale, V.M. et al. The mechanism of mRNA cap recognition. Nature (2024). https://doi.org/10.1038/s41586-024-08304-0
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








#mRNA帽端# #翻译起始#
0