Cell:革命性单细胞研究工具CellHint,可自动化整合多种人类单细胞图谱数据集
2024-01-25 测序中国 测序中国 发表于陕西省
研究团队报道了最新开发的变革性工具—CellHint,这是一种基于预测聚类树(PCT)的工具,可解决数据集之间在注释分辨率和技术偏差方面的细胞类型差异。
导读
在过去的十年里,单细胞基因组学研究描绘了不同发育阶段的各种组织,积累了大量数据集。通过整合这些资源,人类细胞图谱(HCA)等国际组织已经开始绘制人体标准参考图。但不同的实验室会使用不同的细胞类型定义,从而造成命名模式的不一致,这是整合不同细胞图谱数据集的一大挑战。此外,跨数据集细胞类型的标准化还有其他内在挑战,包括由各种技术混淆引起的细胞质量的变化,跨数据集的共享和新细胞类型的存在。
为应对上述挑战,英国Wellcome Sanger研究所、剑桥大学、欧洲生物信息研究所(EMBL-EBI) 等机构的科研人员合作在Cell发表了题为“Automatic cell-type harmonization and integration across Human Cell Atlas datasets”的文章。研究团队报道了最新开发的变革性工具—CellHint,这是一种基于预测聚类树(PCT)的工具,可解决数据集之间在注释分辨率和技术偏差方面的细胞类型差异;能够准确量化细胞间转录组的相似性,并将细胞类型放入一个关系图中,分层定义共享和独特的细胞亚型。研究团队使用CellHint揭示了8种疾病中健康肺细胞状态与患病肺细胞状态之间尚未被充分探索的关系;并将CellHint应用于来自38个数据集的12种组织,提供了一个包含370万个细胞的跨组织数据库以及多种机器学习模型,用于跨人体组织的自动细胞注释。

文章发表在Cell
主要研究内容
用于细胞类型协调和整合的自动化工作流程
CellHint通过两个主要步骤推断数据集之间细胞类型的关系:预测细胞间距离并总结细胞类型之间的比对。CellHint首先在单个参考数据集中计算细胞和细胞类型之间的转录距离,然后使用PCT(一种多输出回归算法)构建该数据集的聚类树。为减少运行时间并避免过拟合,在自顶向下的PCT归纳过程中,聚类树在细胞分类(即节点)处进行修剪。接下来,将来自数据集中每个细胞根据其表达模式分配给匹配的聚类树分支,并以此为基础,预测该细胞与参考数据集中细胞类型的不同之处。
简而言之,一旦为参考数据集构建了PCT,该信息将用于预测细胞在参考细胞类型间的不相似度概况,从而有效地将查询数据集与参考数据集进行比较。通过使用每个数据集作为参考迭代构建这样的聚类树,CellHint推导出一个全局距离矩阵,可表示所有细胞和细胞类型之间的推断差异。
接下来,研究团队定义了一个数据集核心,将原始细胞类型总结为语义上连接的细胞类型,包括匹配和可分割的细胞类型。通个迭代添加新的数据集,将这个核心逐渐扩大,并重新定义了一个扩展的细胞类型库,即CellHint协调表。进一步,CellHint利用细胞类型关系来监督单细胞数据集成
综上,PCT算法连同其他关键步骤和优化,赋予了CellHint稳健、快速和准确的跨数据集分析性能。
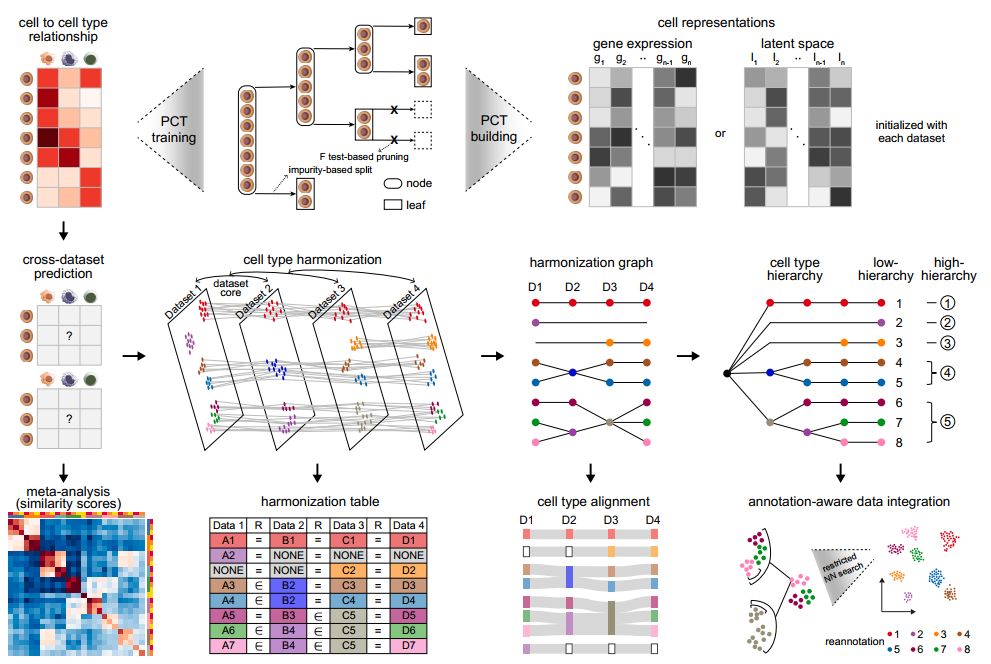
图1. CellHint工作原理和流程
研究团队在5个异质免疫数据集上检测了CellHint的性能,发现CellHint定义的细胞类型与人工注释的细胞类型之间存在一致性,原先需要大量人力来注释、协调的细胞类型在CellHint的算法下都得到了快速的验证。CellHint能够从两个补充角度重建细胞类型关系,其预测了129个原始细胞类型之间的转录组距离,并基于此揭示了高度的跨数据集细胞类型可复制性。除在命名层面上可以确认的协调,CellHint还解决了基于命名难以明显的复杂细胞类型比对。此外,CellHint还能发现先前被忽视的新细胞类型。
总之,通过对多个免疫数据集的专家分级注释进行比较,研究团队展示了CellHint执行整合分析、协调细胞类型、定义细胞层次和提供新的生物学信息的能力。
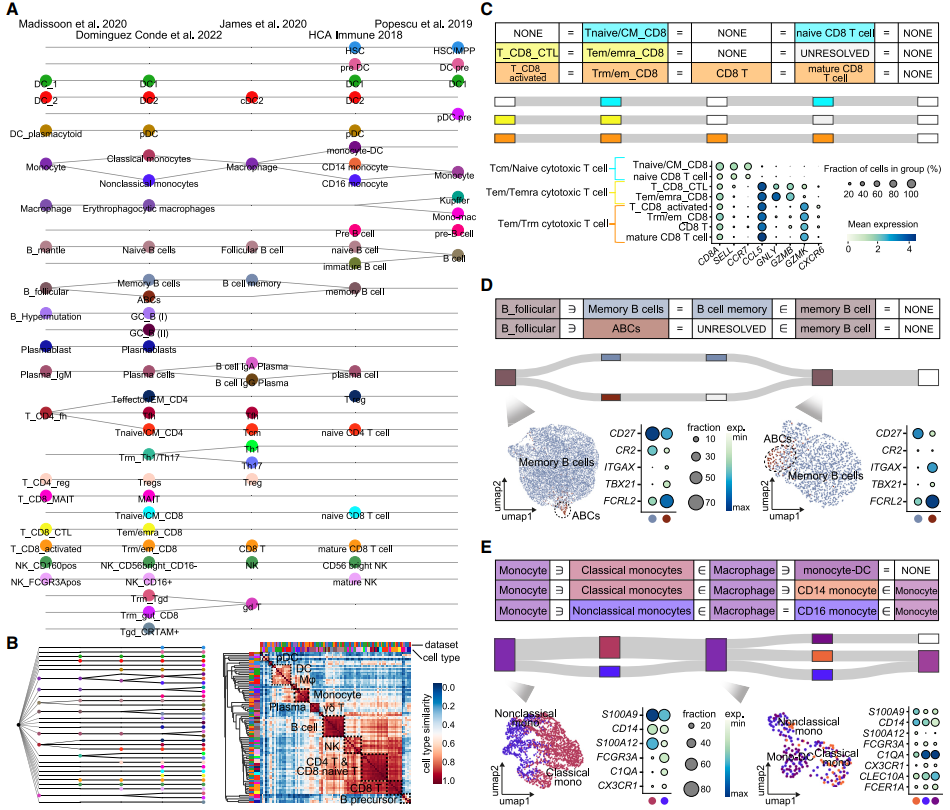
图2. CellHint协调概括了跨越五个免疫数据集的人工注释
解开疾病状态中丰富的细胞状态
在病理条件下,新的细胞类型的出现或相同细胞类型的分子重组,会导致细胞多样性的增加。为检测CellHint在这些情况下的效用,研究团队将CellHint应用于4个成人肺单细胞数据集中,共涵盖8种导致肺纤维化的疾病。这些数据集之间存在明显的技术批次效应,使得发现疾病模式变得具有挑战性。
结果显示,在上述4个数据集中,CellHint协调了不同区域的细胞类型、区分了疾病类型;促进了对转录组变化的全面研究,并解开了多种疾病中的异常细胞状态,揭示了其潜在的病理特征。这些结果表明了CellHint在增强疾病和研究中细胞类型的特异性和可解释性方面的能力。
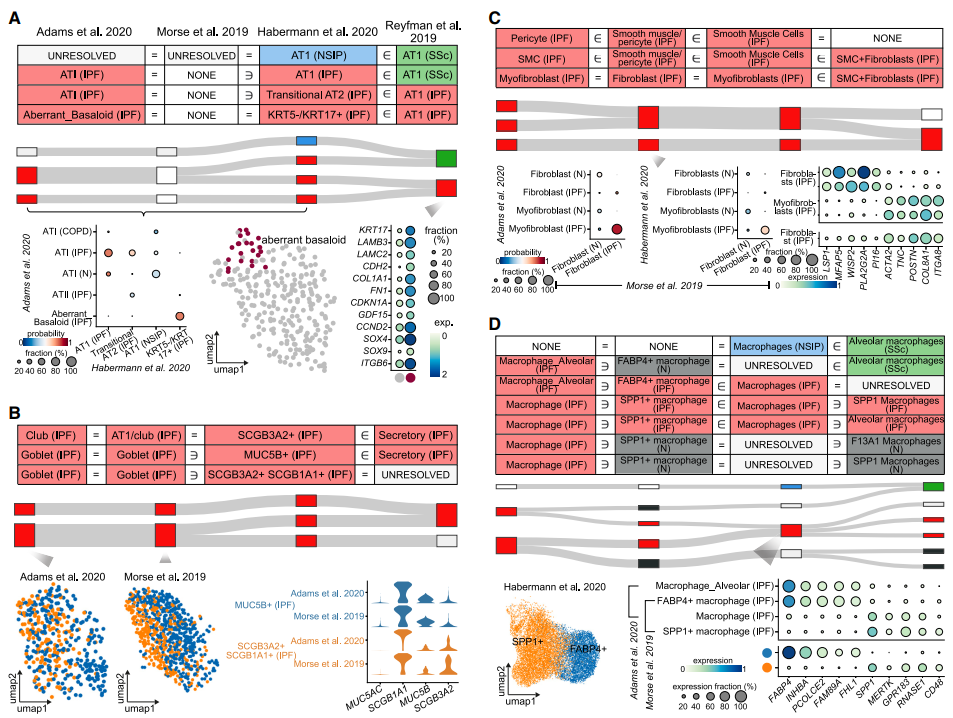
图3. CellHint可用于协调病理细胞状态
通过CellHint进行细胞重新注释和数据整合
根据跨数据集的细胞类型协调,CellHint可以通过在多个数据集中重新注释每个细胞来进一步细化其身份。CellHint将来自5个免疫数据集的细胞重新注释为66个低层次和52个高层次的细胞类型,并为每个细胞提供了不同粒度的精确分类。通过重新注释,所有细胞都被放置在一个共同的命名模式下,使得数据整合可以通过注释进行监督。研究团队采用这一策略整合了来自5个免疫数据集的细胞,并在此基础上揭示了免疫细胞的预期转录组组织。
此外,研究团队在免疫数据集中比较了CellHint与广泛使用的数据整合方法(包括6种无监督方法及2种有监督方法)。结果显示,CellHint能缓解批次效应,并维护跨数据集的细胞类型关系。
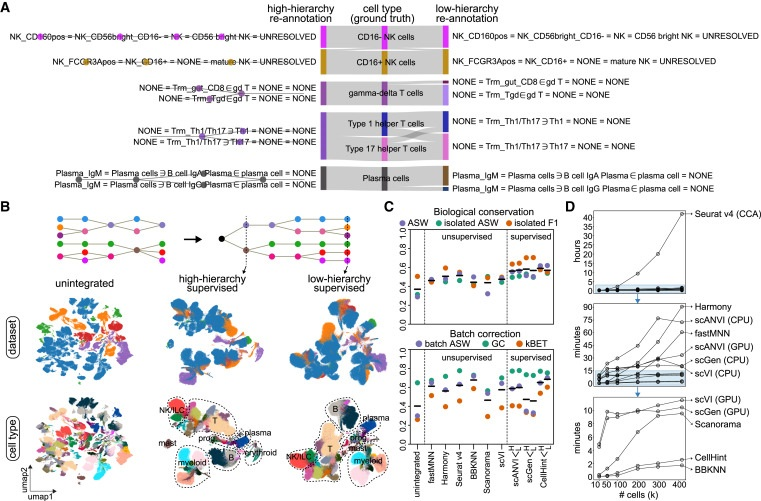
图4. CellHint支持重新注释细胞类型和监督数据集成
细胞协调、整合和分类的多器官参考图
整合单细胞资源是HCA联盟目前关注的重点,但往往费时费力。利用CellHint从协调到整合的高度自动化工作流程,可加速这个过程。因此,研究团队使用CellHint构建了多器官参考图谱。
研究团队共汇集了38个单细胞和单核转录组学数据集,通过CellHint分析了来自369名成人捐献者12个组织、器官的3,694,864个细胞,包括血液、骨髓、心脏、肠、肾脏等,生成了12个标准化的器官参考图谱。这为细胞类型协调、整合和分类等提供了一个可同时检查和查询的多器官资源,可提高器官—器官和健康—疾病的细胞关系的可解释性。
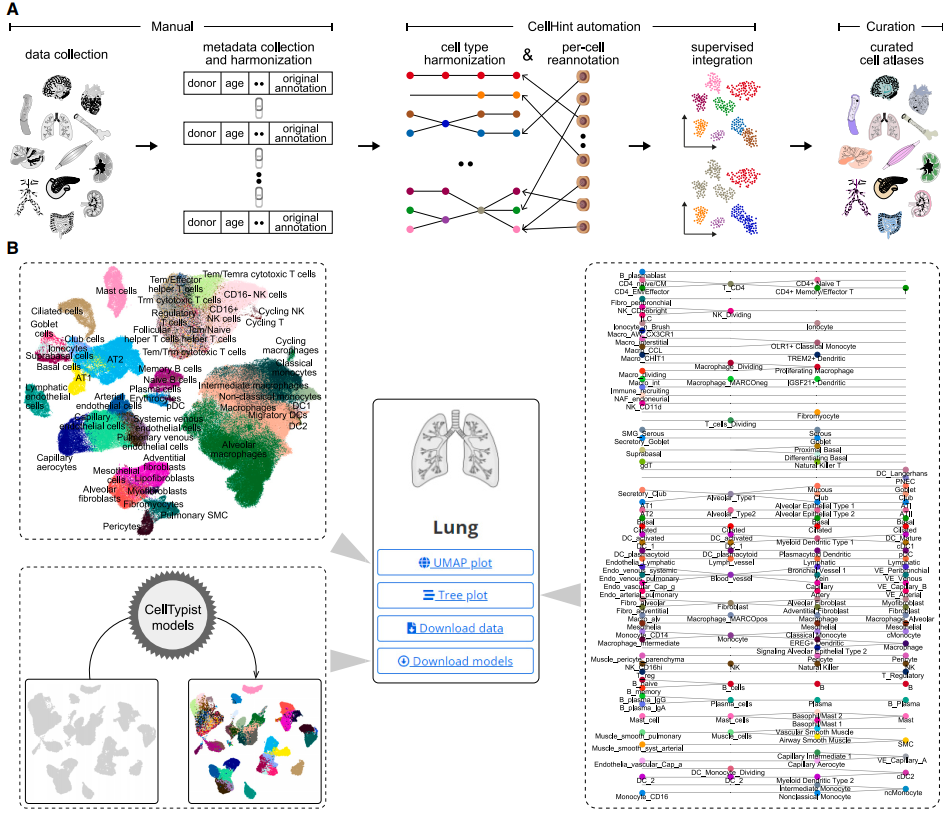
图5. 细胞类型协调、整合和分类的标准化多器官资源
结 语
综上所述,为从跨数据集整合的注释中获益,研究团队开发了用于细胞类型协调和集成的工作流程CellHint,通过评估细胞间的相似性和协调细胞注释来高效地比对多个数据集。研究团队已将CellHint应用于49个数据集,确认了其在数据协调和整合方面的有效性,并提供了一系列器官图谱和机器学习模型,用于自动细胞类型注释。据悉,CellHint在全球范围内都是免费的,其代表了在细胞水平上破译人体复杂性的重要一步,有望改变我们对人类健康和疾病的理解。
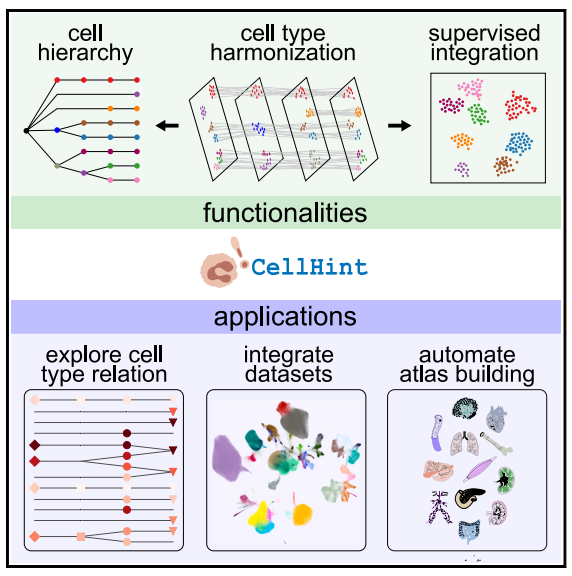
图:总研究概括图
文章第一作者、Wellcome Sanger研究所许川博士表示:“CellHint充分利用了来自独立研究的不一致但有价值的细胞注释信息,以实现生物学驱动的数据整合。有了CellHint,来自独立实验室的细胞可以被重新注释。我们希望这个工具能够极大地促进分子和细胞数据信息在实验室中的再利用。”
文章通讯作者、HCA计划联合发起Sarah Teichmann教授表示:“CellHint实现了单细胞数据的统一和共享,这使得全球研究界能够为世界各地正在进行的研究做出贡献并从中受益,最终有助于推动健康和医疗保健的进步。”
参考文献
Chuan Xu et al, Automatic cell-type harmonization and integration across Human Cell Atlas datasets, Cell (2023). DOI: 10.1016/j.cell.2023.11.026.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








# CellHint# #单细胞图谱#
76