Science:全面揭秘生命密码!基因组基础模型Evo实现分子到基因组水平的序列建模与设计
2024-12-14 测序中国 测序中国 发表于陕西省
研究团队介绍了多模态基因组基础模型Evo,可大规模注释和生成基因组序列。
生命的基本指令编码在生物体的DNA序列中,分析这些指令可以更深入地了解生物学过程,并能以新的方式将生物学重新编程为有用的技术。然而,即使是最简单的微生物基因组也非常复杂,数百万个DNA碱基对编码DNA、RNA和蛋白质的相互作用(分子生物学中心法则),也是细胞功能的关键要素。这种复杂性存在于从单分子到整个基因组的多个维度。
人工智能(AI)的快速发展催生了大语言模型,在对大量数据进行训练时,这些模型表现出越来越先进的多任务推理和生成能力。但这些模型架构的技术局限性限制了其以类似的规模应用于生物学。当前的方法难以在单碱基水平上分析序列,并且只能解释和预测相对较短的 DNA 片段。在大型基因组序列中实现单核苷酸分辨率的先进模型,有可能提取出自然进化变异模式中嵌入的复杂分子相互作用的功能信息。
美国斯坦福大学团队及Arc研究所团队在Science发表了封面文章“Sequence modeling and design from molecular to genome scale with Evo”。研究团队介绍了多模态基因组基础模型Evo,可大规模注释和生成基因组序列。Evo架构利用了深度学习技术,能够高效地处理长序列。通过分析数百万个微生物基因组,Evo模型实现了对生命遗传密码从单个DNA碱基到整个基因组的全面了解。因此,该模型能够解码自然基因组,预测微小DNA变化如何影响生物体的适应性,实现了跨DNA、RNA和蛋白质的预测和设计任务,并在全基因组规模上生成DNA,包括合成CRISPR系统和IS200/IS605转座子。在理解和设计跨模态及多复杂度的生物学方面,Evo实现了重大进步。

文章发表在Science
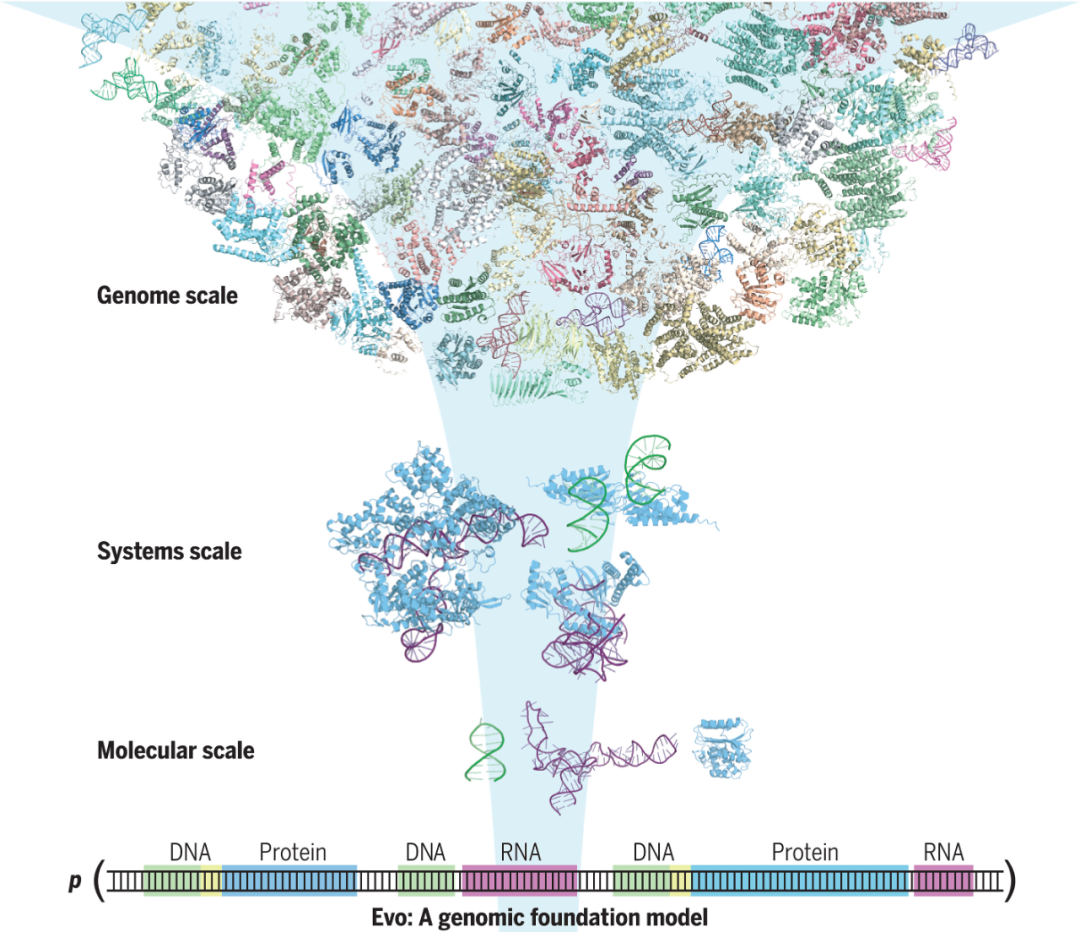
图1. Evo是一个涵盖70亿参数的基因组基础模型。
1 建立核苷酸分辨率的长序列模型
为了高效地建立核苷酸分辨率的长序列模型,研究人员利用了基于深度信号处理的先进架构StripedHyena(图2B),将Evo扩展到70亿个参数,单核苷酸分辨率下上下文长度为131kb。Evo是由29层数据控制卷积算子(Hyena层)与3层(10%)配备旋转式位置编码(RoPE)的多头注意力机制(Multi-Head Attention)交错而成的混合模型。
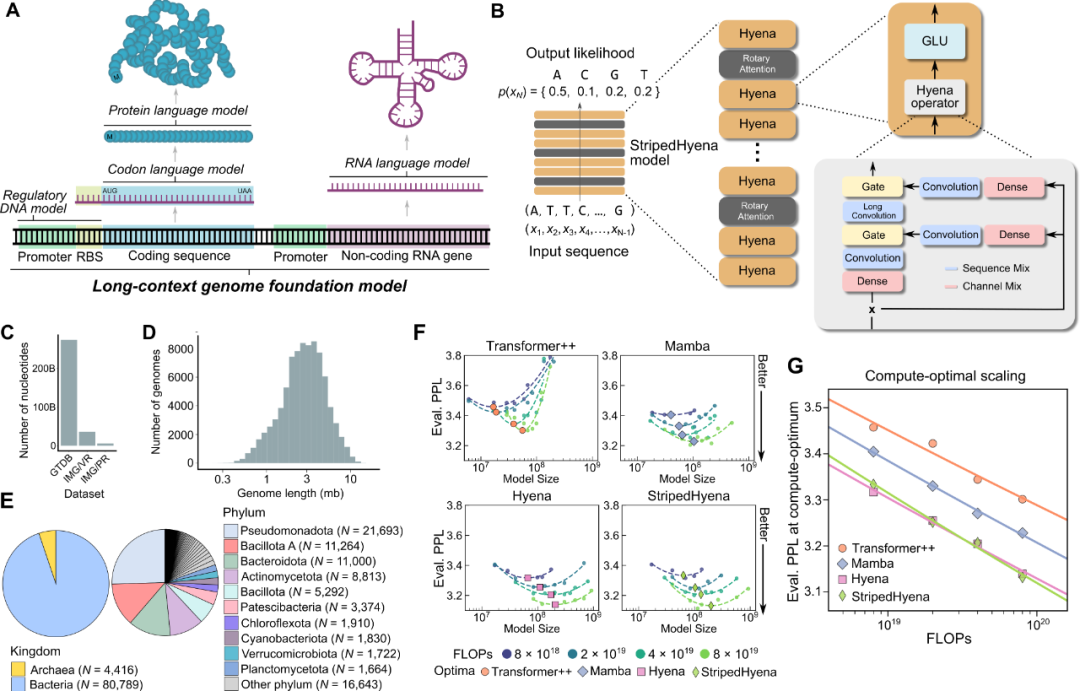
图2. 在原核生命中预训练基因组基础模型。
2 在OpenGenome上大规模训练Evo
研究人员编制了大型基因组数据集OpenGenome,其中包含8万多个细菌和古细菌基因组等数百万个预测的原核生物和噬菌体序列,涵盖3000亿个核苷酸Token(图2C)。预训练包括两个阶段:第一阶段使用8千Token的上下文长度,第二阶段的上下文扩展阶段则使用13.1万Token。
3 在DNA序列数据上展现出良好的尺度定律
研究人员通过计算最优协议比较了不同类别的架构,旨在评估计算最优前沿的结果。在四种架构中训练了300多个模型:Transformer++、Mamba、Hyena和StripedHyena,发现状态空间和深度信号处理架构在Hyena和StripedHyena的缩放率最高。在尺度分析(Scaling Analysis)期间,观察到StripedHyena在所有研究的模型大小和学习率下都可以进行稳定的训练。
4 在DNA、RNA和蛋白质模式中进行零样本功能预测
当使用大肠杆菌蛋白质的深度突变扫描(DMS)数据集评估Evo预测突变对蛋白质功能影响的零样本能力时,发现它优于所有其他测试的核苷酸模型(图3B)。同样,研究人员还评估了Evo使用实验性ncRNA-DMS研究结果作为真实分数进行零样本ncRNA适应度预测的能力(图3C),发现Evo再次优于所有其他测试的核苷酸语言模型。
总之,Evo在没有明确序列注释的情况下通过接受长基因组序列的训练,展示了对组成性蛋白质编码序列、ncRNA序列和调控元件的理解。
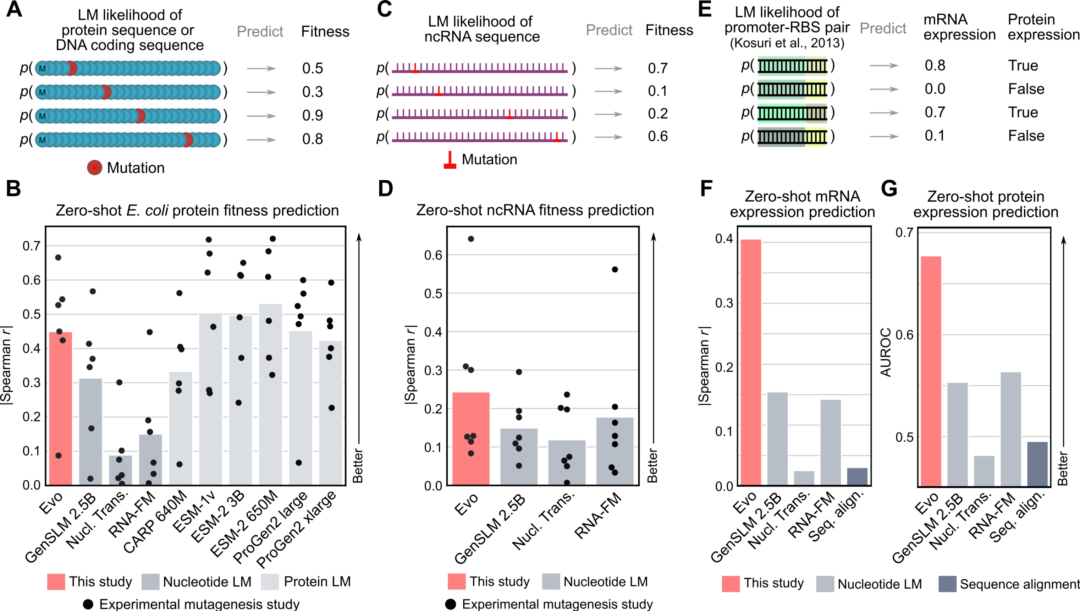
图3. Evo对蛋白质、非编码RNA和调节DNA进行零样本功能预测。
5 CRISPR-Cas分子复合物的生成设计
CRISPR阵列的转录产生非编码CRISPR RNA(crRNA)分子,这些分子与Cas蛋白结合,产生序列特异性DNA靶向所需的功能性防御复合物(图4A)。当对CRISPR-Cas系统进行微调时,Evo可以连贯地生成在序列和结构上与天然存在的Cas系统相似的各种样本。设计新的Cas系统历来依赖于挖掘同源蛋白质的序列数据库,这种方式依赖自然进化来提供功能多样性。而Evo的生成建模提供了一种替代方法,可以在CRISPR技术的广泛应用中加以利用。
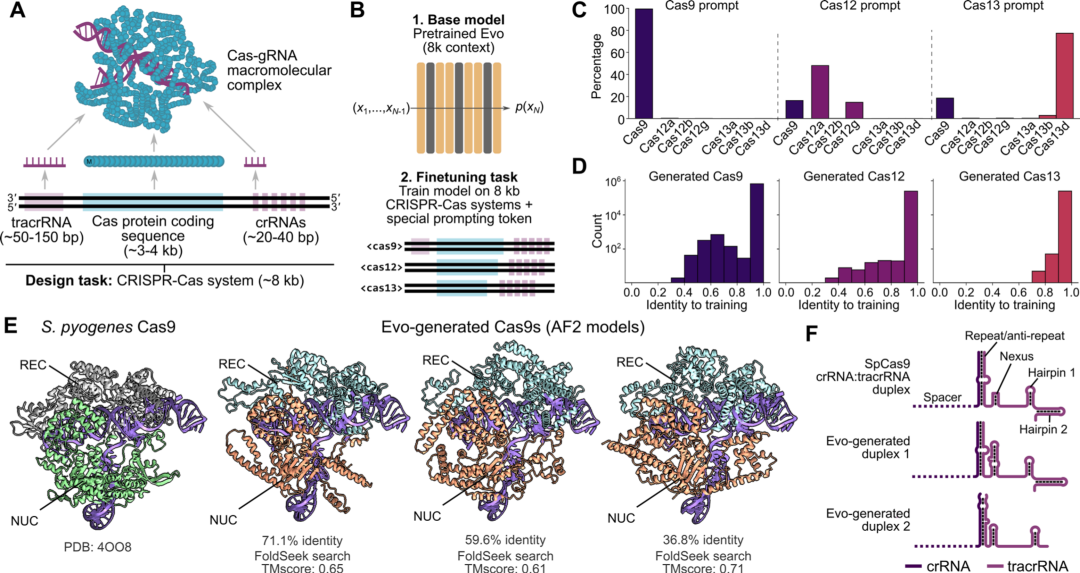
图4. CRISPR-Cas序列的微调使蛋白质-RNA复合物的生成设计成为可能。
6 转座生物系统的生成设计
除了分子复合物,Evo还可以学习多基因系统背后的模式。研究人员在10,720个IS605元件和219,867个IS200元件的自然序列上下文中对Evo进行了微调,并使用该模型生成了新的IS200/IS605元件(图5B)。结果发现,微调模型可以生成具有连贯蛋白质和RNA序列的不同IS605系统,并且Evo正在学习这些元件的重要特征,这些特征可以重新用于改进功能注释。这是蛋白质- RNA和蛋白质-DNA与语言模型共同设计的第一个例子。
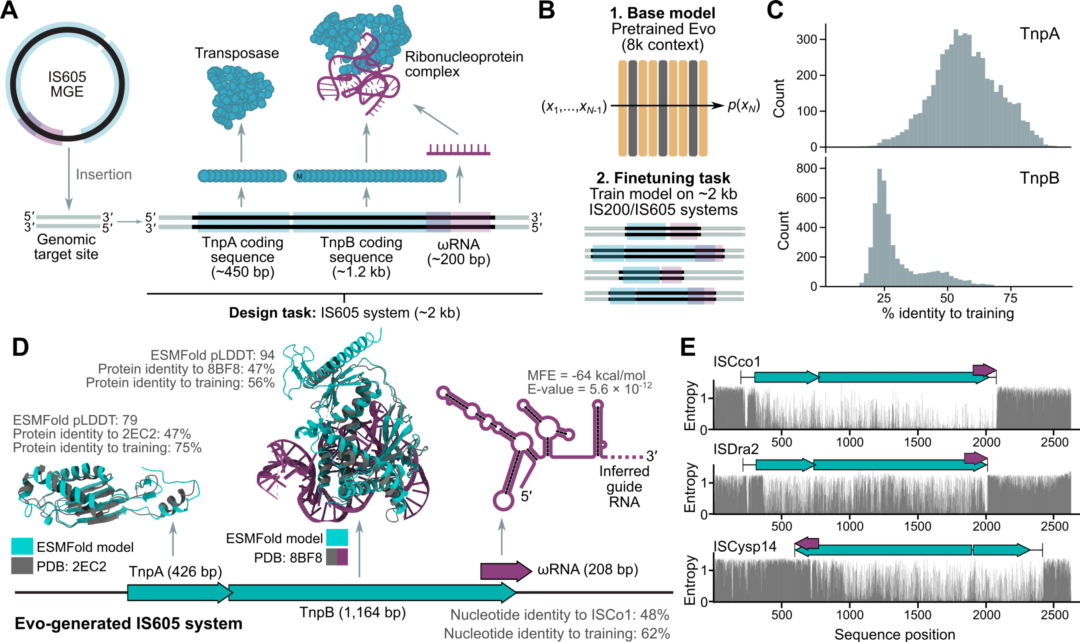
图5. 对IS200/IS605序列的微调使转座生物系统的生成设计成为可能。
7 利用长序列上下文预测基因功能
研究人员以8千预训练Evo模型作为基础模型,在13.1万个Token的序列上进行了第二阶段的预训练(图6A),并预先添加了物种级的特殊Token。结果表明,Evo可以预测许多细菌和噬菌体物种在整个生物体水平上的突变效应,而无需任何明确的基因组注释、特定任务的训练数据或功能标签。与蛋白质或密码子语言模型相比,Evo能够利用从整个基因组中获得的信息,了解核苷酸序列的微小变化如何影响整个生物体的适应性,在更广泛的基因组背景下理解基因功能。
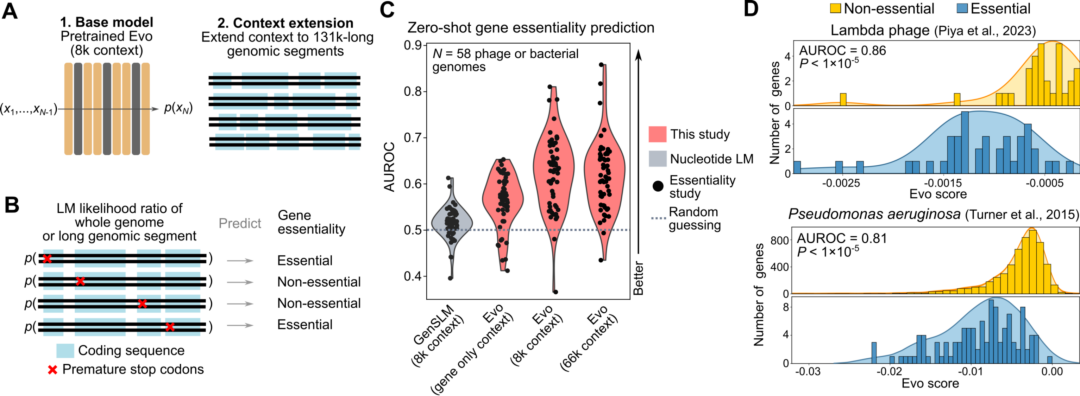
图6. Evo对不同细菌和噬菌体基因组进行零样本基因重要性预测。
8 生成基因组规模的DNA序列
研究人员在不进行额外微调的情况下,在长序列长度下测试了Evo的序列生成质量。这样可以更好地理解模型学习的模式和细节程度,有助于确定模型的能力和局限性。结果显示,Evo可以生成长度超过1Mb可信基因组结构的DNA序列。Evo生成序列的平均编码密度几乎与自然基因组的序列编码密度一样高,并且远高于随机序列的编码密度(图7B)。经过可视化后,自然序列和生成序列都显示出类似的编码组织模式(图7C)。总之,Evo能以前所未有的规模生成包含合理高水平基因组组织的基因组序列,而无需进行密集的提示工程或微调。
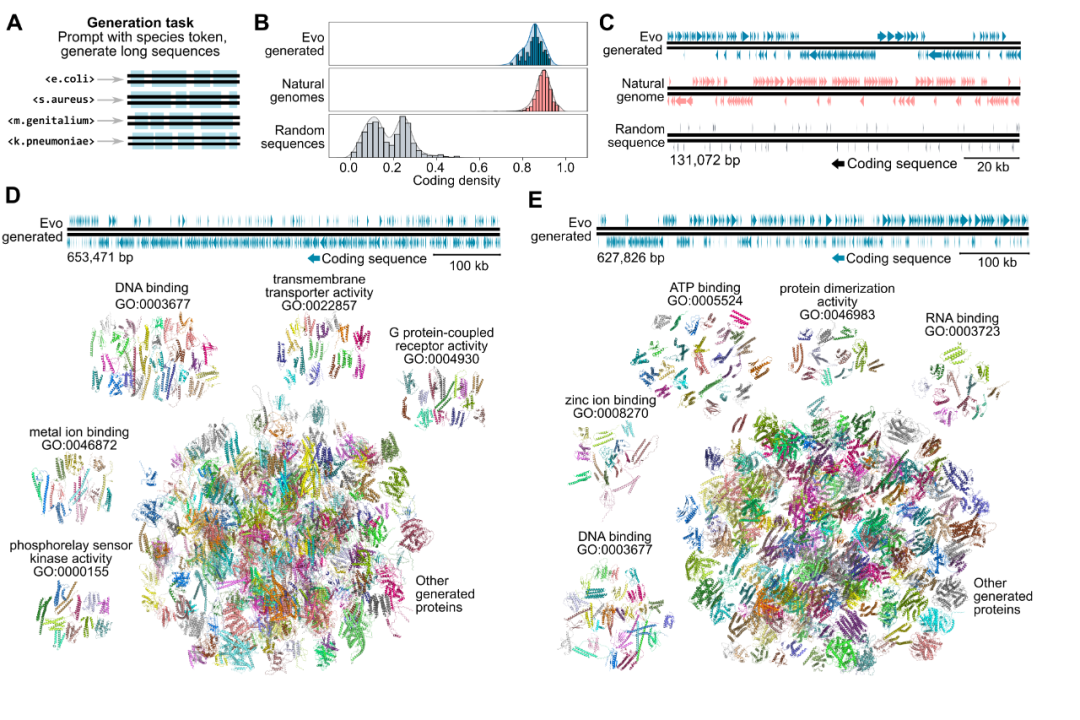
图7. Evo生成具有密集编码结构的基因组级序列。
综上所述,基因组基础模型Evo捕捉了生物学的两个基本方面:中心法则的多模态性和进化的多尺度性。中心法则通过统一的编码和可预测的信息流整合了DNA、RNA和蛋白质,进化则统一了分子、通路、细胞和生物体所代表的跨尺度生物功能。Evo从数百万生物的全基因组序列中学习了这两个方面,从而实现了从分子到基因组尺度的预测和设计。Evo等大规模生物序列模型的进一步发展,与DNA合成和基因组工程的进步相结合,将促进了我们对生物学的理解和控制。
论文原文:
https://doi.org/10.1126/science.ado9336
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









与蛋白质或密码子语言模型相比,Evo能够利用从整个基因组中获得的信息
2
#基因组序列# #多模态基因组基础模型#
12