ISME Journal:中科院深圳先进院戴磊团队解析膳食纤维干预下肠道微生物组的生态应答
2022-05-23 王聪 “生物世界”公众号
基于肠道微生物组的时序数据,解析膳食纤维调控下肠道菌群的动态变化,为系统性理解肠道微生物组的生态学规律以及开发相应的精准营养调控手段奠定了重要基础。团队成员刘红宾博士和美国纪念斯隆-凯特琳癌症中心廖辰
2022年5月21日,中国科学院深圳先进技术研究院合成微生物组学研究中心、深圳合成生物学创新研究院戴磊课题组在 ISME Journal 上发表题为:Ecological dynamics of the gut microbiome in response to dietary fiber的研究论文。
研究团队基于肠道微生物组的时序数据,解析膳食纤维调控下肠道菌群的动态变化,为系统性理解肠道微生物组的生态学规律以及开发相应的精准营养调控手段奠定了重要基础。团队成员刘红宾博士和美国纪念斯隆-凯特琳癌症中心廖辰博士为共同第一作者。

膳食纤维对人体健康有着深远的影响,高膳食纤维摄入量可降低肥胖、炎症性肠病、结直肠癌等多种疾病的风险。越来越多的证据表明,对肠道菌群的调控是膳食纤维发挥其对人体健康益处的重要途径。
值得注意的是,肠道菌群并不是许多各自独立的微生物凑在一起, 它是一个在资源竞争、营养共生和群体感应等相互作用机制下形成的复杂生态系统。因此,在复杂环境中对特定微生物进行调节,通常会牵动整个生态系统,菌群微生物的应答变化很大程度上取决于微生物之间存在的竞争、互惠等生态互作关系(图1)。
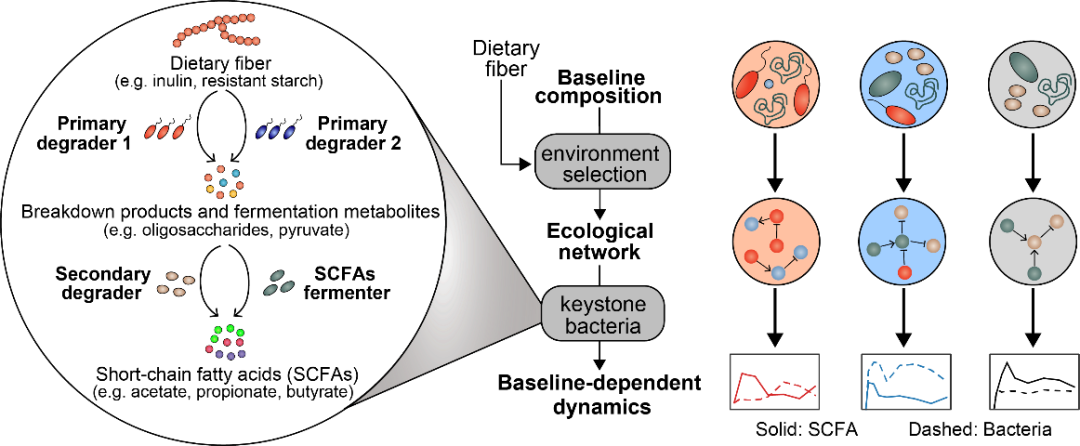
图1:微生物生态对肠道菌群应答的影响
研究团队通过生态动力学模型,解析膳食纤维干预下肠道菌群应答的生态学机制。以广义Lotka-Volterra (gLV) 模型为框架,拟合小鼠肠道菌群在膳食纤维干预后的动态变化,推断膳食纤维干预下生长速率有显着响应的关键微生物和不同微生物之间的互作关系。通过这一方法,该团队成功推断了小鼠肠道菌群中膳食纤维代谢功能的关键响应菌,以及关键微生物之间的互作关系。当关键微生物缺失时,膳食纤维对肠道菌群的调控作用可能会“失效”(图2)。
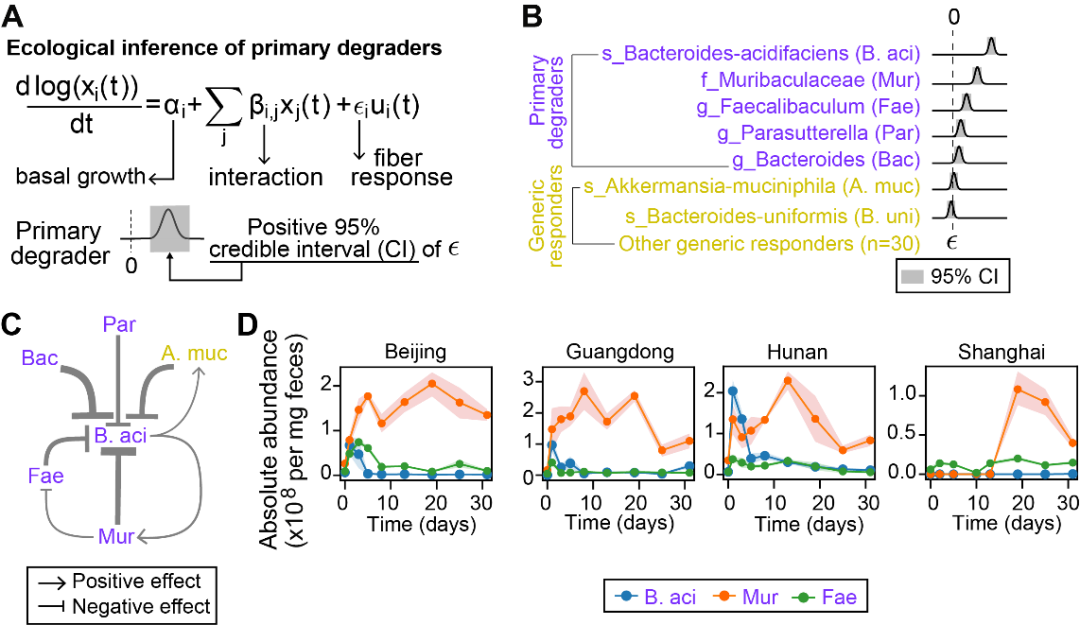
图2:利用生态动力学模型解析小鼠肠道菌群对菊粉干预的响应
研究团队进一步将生态学模型用于分析人体肠道微生物组在膳食纤维干预下的时序数据,成功推断出具有菊粉代谢功能的青春双歧杆菌。此外,发现了青春双歧杆菌与粪便拟杆菌之间存在的竞争关系,并通过体外培养实验进行了验证。比如,通过向肠道菌群中补充青春双歧杆菌,可以观察到粪便拟杆菌丰度的下降(图3)。
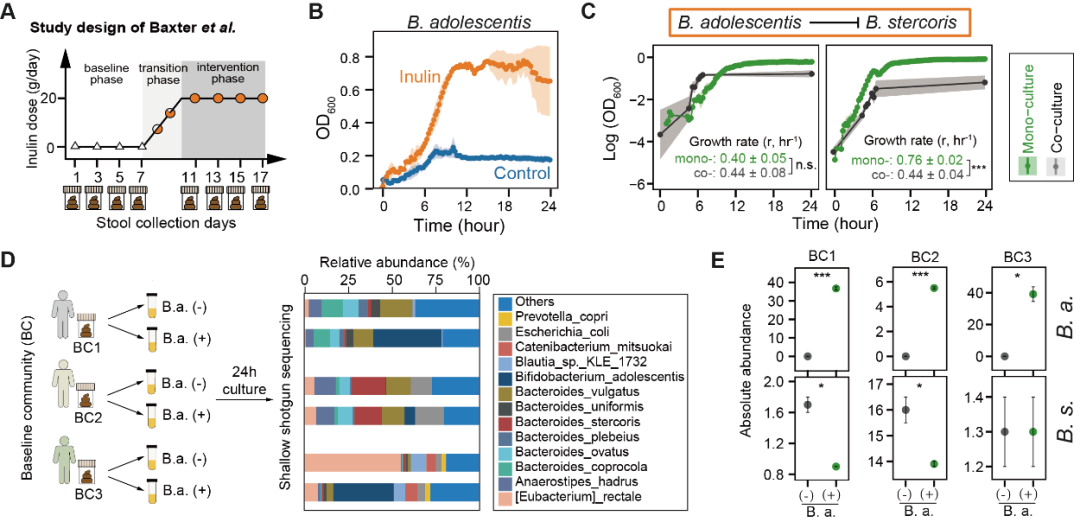
图3:人体肠道菌群在菊粉干预下的关键菌和互作验证
针对膳食纤维干预下肠道菌群的个性化应答现象,研究结合时序数据与数学模型,深入解析肠道菌群的生态学规律。研究揭示了微生物生态网络对于理解与预测肠道菌群应答的重要性,为开发生态层次的靶向调控手段奠定了理论基础。
PI与课题组简介:
戴磊博士,研究员,中科院深圳先进技术研究院合成生物学研究所,合成微生物组学研究中心主任。国家重点研发计划青年项目负责人,入选《麻省理工科技评论》中国区“35岁以下科技创新35人”。
戴磊课题组的主要研究方向为宿主共生微生物组的生态和进化。运用定量生物学、合成生物学的工具,对微生物组的结构和功能进行精准解析和调控,致力于解决人体健康、农业生产等重大问题。
拟招聘具有微生物学、合成生物学、生物信息学等相关研究背景博士后1-2名,客座学生1-2名。有意申请者请将个人简历以邮件方式发送至 lei.dai@siat.ac.cn。
原始出处:
Liu, H., Liao, C., Wu, L. et al. Ecological dynamics of the gut microbiome in response to dietary fiber. ISME J (2022). https://doi.org/10.1038/s41396-022-01253-4.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#中科院#
128
#解析#
114
#微生物#
85
#膳食#
120
#微生物组#
106
#肠道微生物组#
111