Nat Commun:序列末端依赖性PCR方法——STEM-PCR,以单碱基分辨率识别位点特异性突变
2023-03-31 小鹿-先森 测序中国 发表于上海
研究团队展示了一种简单通用的PCR策略来识别核酸修饰和单碱基突变。该方法被命名为特异性末端介导PCR,具有高特异性,并且能够以单碱基分辨率识别任何序列的位点特异性变异。
核酸位点特异性突变已被证实可以作为疾病的临床生物标志物,包括癌症、神经系统疾病等。单碱基分辨率核酸分析新方法的发展,如纳米孔和单分子实时测序方法,有助于检测核酸位点特异性变化,包括单碱基突变。但这些方案实施起来相对复杂,限制了其在临床中的应用。
目前,聚合酶链式反应(PCR)被整合到许多常规临床分析工作流程中。但目前这些方法还不能在单碱基水平上识别位点特异性突变,也不能检测表观遗传碱基修饰,使得多个研究团队开发了增强型或改良型PCR方案。
近日,来自上海交通大学生物医学工程学院/Med-X研究院的研究团队在Nature Communications发表了题为“Sequence terminus dependent PCR for sitespecific mutation and modification detection”的文章。研究团队展示了一种简单通用的PCR策略来识别核酸修饰和单碱基突变。该方法被命名为特异性末端介导PCR(Specific Terminal Mediated Polymerase Chain Reaction, STEM-PCR),具有高特异性,并且能够以单碱基分辨率识别任何序列的位点特异性变异。

文章发表在Nature Communications上
科学与技术
1 STEM-PCR的设计理念和优化
STEM-PCR依赖于对目标序列的简单处理,从而产生一个携带特定末端部分的分子结构,并具有自折叠和启动PCR引物结合进行扩增的能力。因此,该技术只需要一个简单的DNA序列工具箱和形成复合物的特定设计机制。
研究团队首先展示了利用限制性内切酶(RE)消解的STEM-PCR策略进行位点特异性检测的基本概念。当目标序列存在时,酶消化产生具有特定5 '末端的序列P1,未消化的目标保持完整。在扩增步骤中,P1作为模板,通过定制设计的可折叠引物(TFP)启动线性链合成,该引物与已消化和未消化的模板杂交。对于被消化的分子P1,这一过程止于5 '端,生成一个可以自折叠(P2)和自启动的结构,形成一个完整的发夹结构(P3),没有3 '端突出。对于未消化的分子,继续合成,防止自启动(P4)。完整的发夹结构(P3)使用人造引物(Artificial primer, AP)启动指数扩增过程。
与传统PCR扩增相比,STEM-PCR识别序列特异性信息能力的创新,是通过处理模板和TFP的不同序列成分设计的中间结构的生成。折叠区域(FR)的长度对稳定性的影响可转化为更高的放大效率。
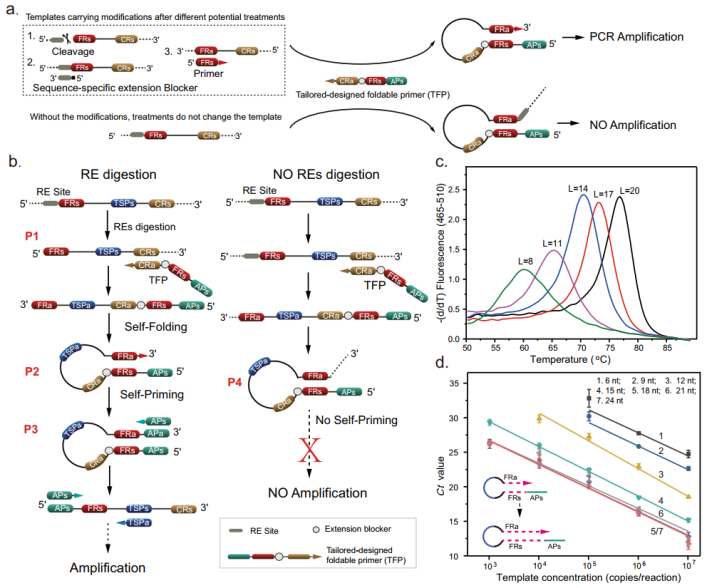
图1. STEM-PCR设计原理图,来源:Nature Communications
2 STEM-PCR检测单位点甲基化
为了验证STEM-PCR策略,研究人员将DNA甲基化和突变作为具有重要实际临床意义的案例研究,并设计了三种具有高临床相关性的测定方法来检测DNA甲基化,包括同时检测多个位点的甲基化。
验证结果显示,STEM-PCR与未甲基化序列无交叉反应,且没有敏感性损失。STEM-PCR产物对应的条带进行测序,发现末端存在AP和TSP,表明STEM-PCR的机制和反应步骤符合预期。
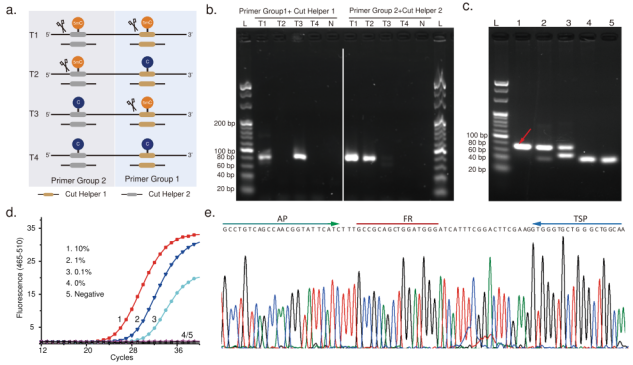
图2. STEM-PCR有效检测单位点甲基化,来源:Nature Communications
3 利用STEM-PCR检测单碱基突变
研究人员使用STEM-PCR检测了EGFR中的L858R单碱基突变,发现该方法表现出与酶法相似的高性能,即使在3000拷贝/反应的浓度下,也具有30拷贝/反应的敏感性和高特异性。
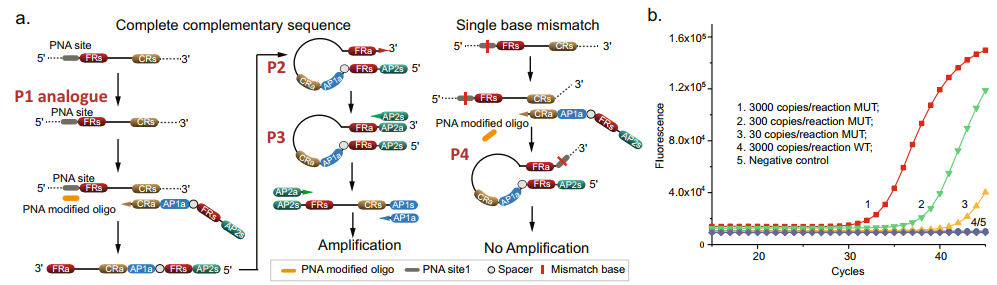
图3. STEM-PCR有效检测单碱基突变,来源:Nature Communications
4 STEM-PCR检测共甲基化
为证明STEM-PCR可以很容易地用于检测共甲基化,研究人员从培养细胞中提取人类基因组DNA,并将其碎片化到约150 bp作为ctDNA甲基化的模型,共甲基化分析结果显示了该方法的特异性,并通过反应产物测序得到了证实。
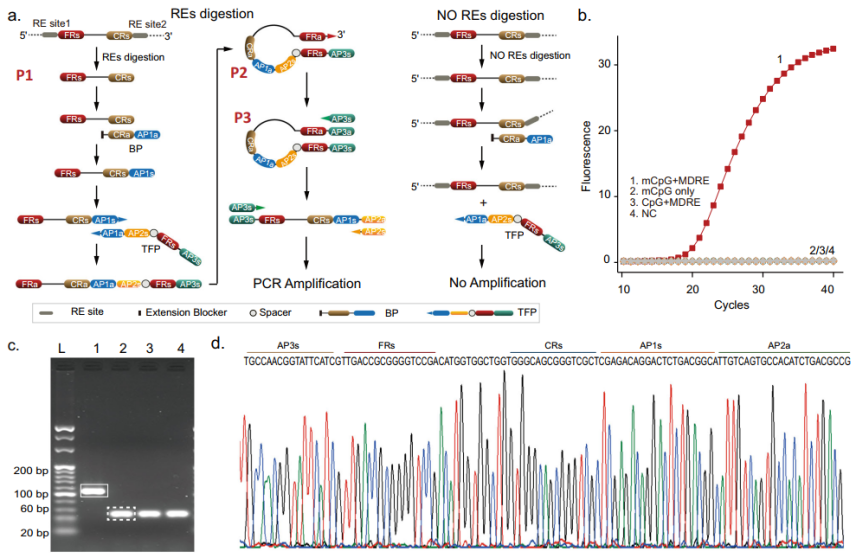
图4. STEM-PCR检测共甲基化的能力,来源:Nature Communications
结 语
遗传变异的检测促进了用于诊断目的的方法和分析的发展。但每种方法都有其自身的缺点,包括敏感性、特异性和/或所需的冗长处理。相比之下,该研究提出的STEM-PCR是一种简单有效的策略,是一种可检测不同修饰且值得推广的方法。该方法基于中间二级核酸结构的生成,使用一组引物序列与目标序列特异性相互作用和杂交,生成具有明确末端结构的构建物,可以自我折叠成发夹结构和自我启动用于PCR扩增。
研究人员通过多种方式证明了该方法的潜力,该方法不使用亚硫酸氢盐制备,而是使用酶消化,以检测单个或多个甲基化位点,以及用于无酶测定。同时,该技术可以很容易地扩展到使用基因编辑工具的其他方法,例如CRISPR。
参考文献:
1. Xu, G., Yang, H., Qiu, J. et al. Sequence terminus dependent PCR for site-specific mutation and modification detection. Nat Commun 14, 1169 (2023).
2. Tse, O. O. et al. Genome-wide detection of cytosine methylation by single molecule real-time sequencing. Proc. Natl Acad. Sci. USA 118, e2017421118 (2021).
3. Widschwendter, M. et al. Epigenome-based cancer risk prediction: rationale, opportunities and challenges. Nat. Rev. Clin. Oncol. 15, 292–309 (2018).
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言







