Genome Biology:纳米孔测序可用于检测cfDNA的细胞起源和癌症特异性甲基化特征
2022-08-09 测序中国 测序中国
该研究结果表明,基于cfNano和Illumina的WGS和WGBS方法在检测DNA甲基化、片段化和CNA的癌症特异性特征方面性能基本一致。
细胞游离DNA(cfDNA)携带起源组织的信息特征,包括基因组改变、DNA修饰和细胞类型特异性片段化模式等。其中,DNA甲基化是泛癌筛查研究中很有应用前景的cfDNA生物标志物。
近年来,以Oxford Nanopore Technologies(ONT)为代表的纳米孔测序技术日渐成熟,其在DNA甲基化检测的准确性上也已被证实,可以产生高分辨率的单碱基对。便携式的ONT平台不需要复杂的样品处理步骤和PCR扩增,具有低建库成本和快速测序工作流程优势,可实现实时医疗,对临床应用具有非常大的吸引力。ONT另一主要优势在于可获取长读长序列,而捕获更长cfDNA片段有可能发现新的生物标志物。

文章发表在Genome Biology
近日,来自以色列希伯来大学等单位的研究人员在Genome Biology杂志发表了题为“Detecting cell‑of‑origin and cancer‑specific methylation features of cell‑free DNA from Nanopore sequencing”的研究文章。研究团队进行了循环肿瘤DNA(ctDNA)ONT测序的可行性研究,基于ONT全基因组测序检测癌症患者ctDNA的拷贝数变异(CNA),并通过比较ONT测序与Illumina测序平台得获得的甲基化和片段特征信息,证实了ONT测序可成为液体活检的有力工具。
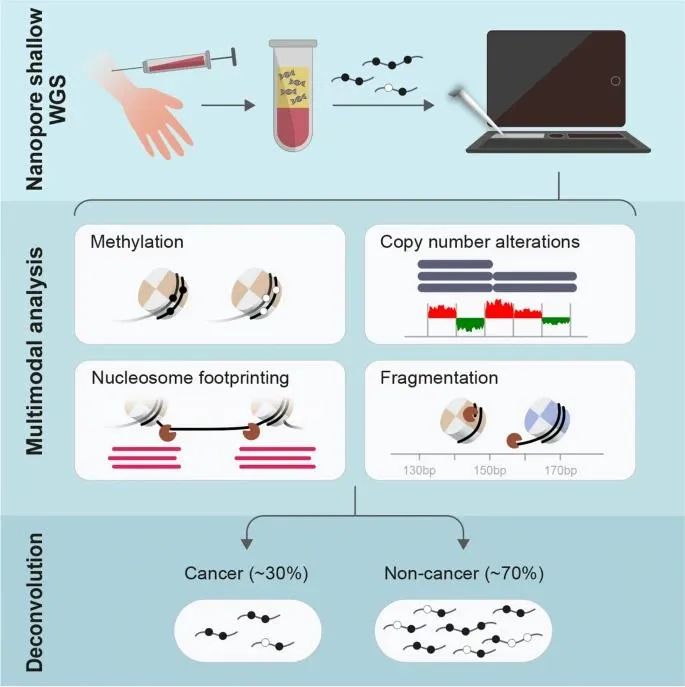
研究概要图
主要研究内容
基于cfNano评估细胞类型
首先,研究人员使用已发表的基于亚硫酸氢盐测序(Illumina平台)的DNA甲基化数据集和基于游离DNA纳米孔测序(cfNano)的DNA甲基化数据对健康血浆cfDNA进行了细胞类型反卷积分析。为了更好地理解cfNano相对较低的测序深度对数据分析结果的影响,研究人员从最低0.0001X的基因组覆盖度开始计算,发现从较高深度覆盖到~ 0.2X,所有样本均以预期的细胞类型为主:单核细胞、淋巴细胞、巨核细胞、中性粒细胞/粒细胞。

图1. 基于cfNano评估细胞类型,来源:Genome Biology
接下来,研究团队使用ichorCNA工具计算了体细胞拷贝数变异(CNAs),以评估cfDNA中癌细胞的比例,即“肿瘤比例”。结果显示,虽然Illumina平台的测序深度明显更高,但cfNano和Illumina测序的肿瘤得分高度相似。研究团队设计了一种基于NNLS回归的“双组分”反卷积方法,发现这些基于DNA甲基化的评分和ichorCNA对癌细胞组分的评分在很大程度上是一致的,6个肺癌患者样本的肿瘤评分均显著增高于7个健康血浆样本的分数。此外,研究人员通过不同的策略验证了甲基化反卷积的鲁棒性。
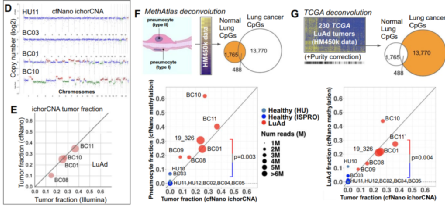
图2. 基于不同测序平台的肿瘤分数评估,来源:Genome Biology
使用cfNano检测DNA甲基化变化的基因组环境
研究团队在已发表的基于Illumina平台的全基因组甲基化测序(WGBS)研究和cfNano研究中分析了NKX2-1位点的血浆cfDNA甲基化水平。在三个WGBS研究的健康样本和cfNano样本中,NKX2-1位点被完全甲基化,疾病组存在显著的去甲基化。
全局DNA低甲基化是癌症表观基因组的特征之一,长期以来一直被认为是ctDNA的通用标记。研究人员重新分析了TCGA健康肺和肺肿瘤组织的WGBS甲基化数据,发现了一个典型的染色体臂,其在肿瘤样本中的部分甲基化区域(PMD)发生强低甲基化。这种强烈的低甲基化也同样只出现在cfNano癌症样本中相同的PMD区域。总的来说,与健康对照相比,低甲基化程度在所有癌症样中明显更高,且这种现象在两种测序平台中均可检测到。
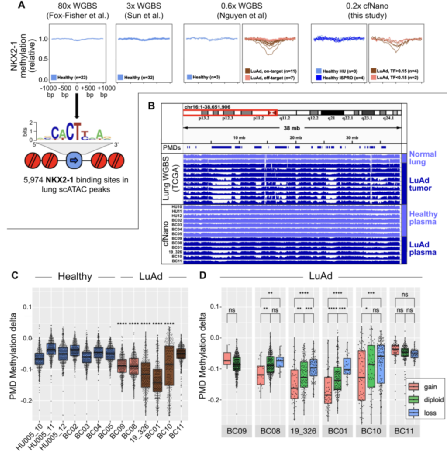
图3. 基于cfNano检测DNA甲基化变化的基因组环境,来源:Genome Biology
cfNano与Illumina WGS检测的癌症相关cfDNA片段长度特征
cfDNA中特定的片段长度与癌症衍生的ctDNA片段有关,并且已经被用作准确的癌症分类工具。特别是较短的单个核小体片段 (< 150 bp) 往往富集了癌症来源的片段。片段长度密度图显示,与健康对照相比,cfNano肿瘤样本富含单个核小体片段。此外,研究团队将cfNano库中的单个核小体片段与基于Illumina WGS文库的片段进行比较,发现两种文库类型具有很强的相关性。有趣的是,这种相关性在健康样本和癌症样本中都存在,表明同样的潜在机制影响来自癌症和非癌症细胞类型的cfDNA。
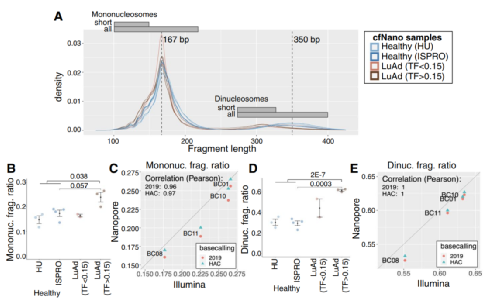
图4. 癌症相关碎片长度特征的比较,来源:Genome Biology
cfNano与Illumina WGS检测的癌症相关cfDNA片段末端特征
前期研究结果表明,cfDNA片段位点紧邻侧翼的4个碱基序列存在偏倚。为了在cfNano样本中研究这一点,研究人员首先绘制了在基于Illumina平台的健康血浆cfDNA研究中发现的25个最丰富的4碱基末端基序,并使用热图来表明每个cfNano样本和匹配的Illumina样本中的基序频率。结果显示,基于两个平台的4碱基末端基序的相对频率总体上是高度一致的。
最后,研究人员聚焦在CCCA末端基序,这通常是健康血浆中最丰富的4碱基末端基序,其减少被证明是几种癌症类型的癌症标志物,包括肺癌。cfNano和Illumina WGS样本中发现,CCCA的频率确实是最高的,在高肿瘤比例的癌症样本中,CCCA的频率明显低于健康样本。
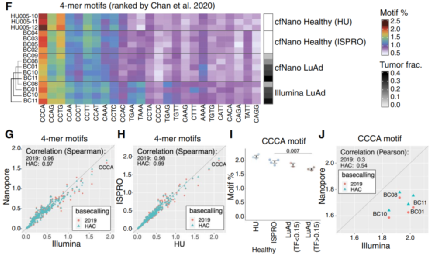
图5. 癌症相关cfDNA片段末端特征比较,来源:Genome Biology
结 语
综上所述,该研究结果表明,基于cfNano和Illumina的WGS和WGBS方法在检测DNA甲基化、片段化和CNA的癌症特异性特征方面性能基本一致。研究还发现,以cfNano为目标的基因组覆盖率最低为0.2X,即可根据DNA甲基化检测所有样本中的癌症衍生DNA。
由于该研究样本量太小,无法确定甲基化检测敏感性和特异性的下限,但细胞类型特异性和癌症特异性甲基化都存在于该研究涉及的大多数样本中,这些结果为更大规模的研究分析提供了支持。
参考文献:
1. Katsman et al. Detecting cell‑of‑origin and cancer‑specific methylation features of cell‑free DNA from Nanopore sequencing. Genome Biology (2022) 23:158.
2. Zemmour H, et al. Non‑invasive detection of human cardiomyo‑cyte death using methylation patterns of circulating DNA. Nat Commun. 2018;9:1443.
3. Liu Y, et al. DNA methylation‑calling tools for Oxford Nanopore sequencing: a survey and human epigenome‑wide evaluation. Genome Biol. 2021;22:295.
·END ·
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









#Bio#
70
#cfDNA#
100
#纳米孔#
94
#特异性#
102
#Biol#
90