Nat Methods:谢晓亮课题组开发单细胞联合三维基因组和转录组测序技术解析嗅觉受体选择机制
2024-04-23 测序中国 测序中国 发表于陕西省
该工作为理解嗅觉受体增强子与嗅觉受体基因的空间互作如何动态协调“一个神经元,一个受体”选择过程提供了宝贵见解。
嗅觉是我们基本的感觉之一,我们能够准确地识别各种的气味分子,这依赖于表达在嗅觉神经元表面的嗅觉受体。在哺乳动物中,嗅觉受体基因构成了最庞大的基因家族,在小鼠基因组中存在超过1000个,在人类基因组中存在超过400个。这些基因以基因簇的形式分布在不同染色体上。尽管嗅觉受体库如此庞大,但每个成熟的嗅觉神经元只会随机地表达一个嗅觉受体的一个等位基因,这一现象被称为“一个神经元,一个受体”
此前系列研究表明,嗅觉受体的表达受到三维基因组的紧密调控,表现为两个方面:一是嗅觉受体簇形成抑制性的空间聚集体,以抑制嗅觉受体的表达;另一方面是嗅觉受体聚集体将位于基因间区域的63个增强子汇聚,从而形成多染色体增强子中心以激活单一嗅觉受体的表达。然而,对于单一嗅觉受体的调控机制仍然存在一些未解决的问题,比如单细胞RNA-seq实验发现嗅觉受体的表达是逐步发生的:祖细胞随机表达一组(5–15)嗅觉受体,随后其中一个嗅觉受体将其他嗅觉受体竞争失活最终成为被选择的嗅觉受体。此前研究的结论都基于成熟嗅觉神经元,此时嗅觉受体选择已经完成,在嗅觉受体逐步表达过程中多染色体增强子如何调控嗅觉受体表达仍不清楚。此外,谢晓亮课题组此前解析了嗅觉神经元的单细胞三维基因组结构,发现每个神经元存在多个增强子聚集体并推测表达的嗅觉受体位于最大的增强子中心【1】。但是此结果表明与增强子的互作本身并不足以实现嗅觉受体的激活。由于嗅觉受体表达的随机性和多样性,此前技术无法解决这些问题。
2024年4月15日,北京大学生物医学前沿创新中心/昌平国家实验室谢晓亮教授及其团队在Nature Methods杂志发表了题为“Simultaneous single-cell three-dimensional genome and gene expression profiling uncovers dynamic enhancer connectivity underlying olfactory receptor choice”的研究论文,揭示了嗅觉受体选择背后的三维基因组调控机制。该工作为理解嗅觉受体增强子与嗅觉受体基因的空间互作如何动态协调“一个神经元,一个受体”选择过程提供了宝贵见解。哥伦比亚大学Stavros Lomvardas在同期发表了对此工作的评论文章【2】。

为了研究此问题,研究人员开发了一种名为LiMCA的单细胞联合三维基因组和转录组多组学测序技术,通过物理核质分离的方法,实现了高检测率全长转录组和高分辨率三维基因组结构的同时检测(图1)。
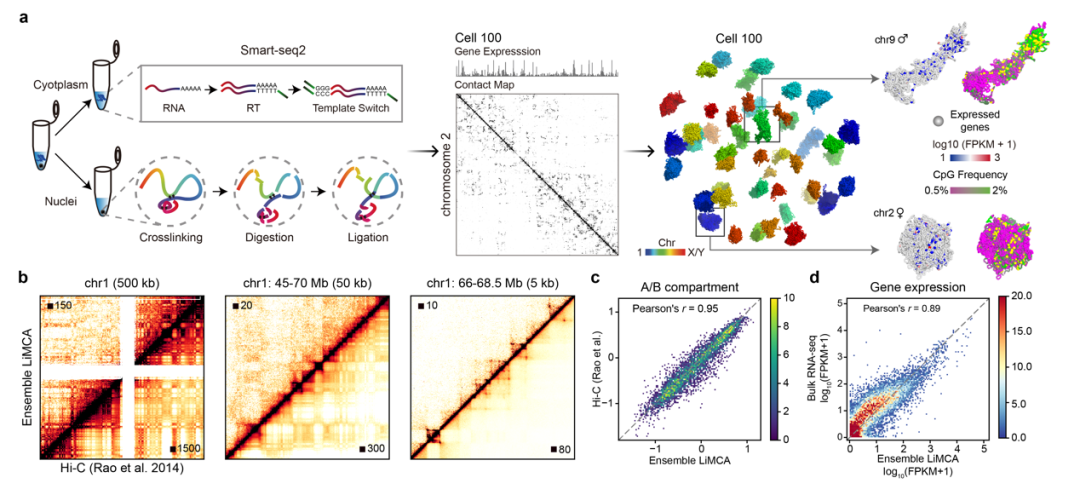
图1. 发展单细胞联合三维基因组和转录组检测技术LiMCA
结合单细胞转录组、单细胞染色质开放性检测技术METATAC【3】和LiMCA技术,研究团队构建了不同发育阶段的嗅觉神经元多组学图谱,以研究嗅觉受体选择的染色质基础(图2a-c)。根据高分辨率染色质开放性图谱,研究团队鉴定到了27个新的嗅觉受体增强子,并发现这些增强子与之前鉴定到的63个增强子具有相似的转录因子结合特征和LHX2/EBF复合基序(图2d-e)。研究团队通过对染色质开放性图谱进行拟时间分析,揭示了嗅觉受体增强子和嗅觉受体基因随嗅觉神经元发育过程中的开放性动态变化:发现增强子和嗅觉受体在祖细胞阶段开放性达到顶峰,随着神经元成熟增强子开放性逐渐下降(图2f)。此结果表明增强子的动态开放协调了嗅觉受体的逐步表达。
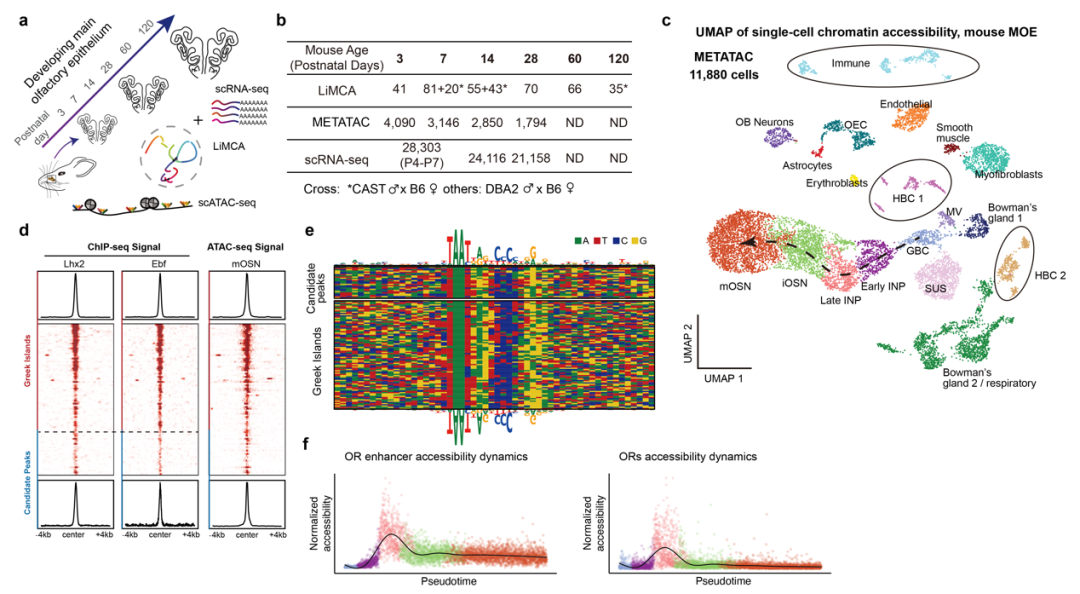
图2. 构建嗅觉神经元多组学图谱,揭示嗅觉受体增强子开放性动态
通过LiMCA多组学图谱,研究团队成功捕获了不同发育阶段的嗅觉神经元(图3a)。基于嗅觉受体表达信息,研究团队将细胞分为多嗅觉受体激活阶段、嗅觉受体沉默阶段和单一嗅觉受体表达阶段(图3b)。LiMCA提供的配对的嗅觉受体表达和三维基因组结构信息,使得研究团队可以直接研究不同嗅觉受体表达阶段活跃的嗅觉受体和增强子的三维空间位置关系。对不同表达阶段的细胞分析后,研究团队发现:在多嗅觉受体激活阶段,来自同一条染色体的顺式增强子(cis-enhancer)就足够激活嗅觉受体表达(图3b-c);在嗅觉受体沉默阶段,占主导地位的嗅觉受体周围增强子数目显著多于低表达的嗅觉受体(正在沉默的嗅觉受体)(图3d-e);在单一嗅觉受体表达阶段,最终被选择的嗅觉受体通常不位于最大的增强子中心(图3f-g)。

图3. LiMCA多组学图谱揭示了嗅觉受体逐步选择背后的增强子空间互作基础
综上,该研究通过开发高灵敏度单细胞同时检测染色质结构和基因表达技术LiMCA,同时结合单细胞染色质开放性检测技术METATAC,揭示了嗅觉受体随嗅觉神经元发育过程中逐步表达背后的染色质开放性和结构基础(图4a)。

图4. 嗅觉受体逐步表达过程中增强子空间互作模型
北京大学生物医学前沿创新中心/生命科学学院博士生吴烘贵、张剑坤,北京大学生物医学前沿创新中心/化学与分子工程学院博士生简繁冲为该论文的并列第一作者。北京大学生物医学前沿创新中心谢晓亮教授和斯坦福大学神经生物学系谭隆志博士为该论文共同通讯作者。北京大学生物医学前沿创新中心郑英慧副研究员为该论文做出了重要贡献。该研究得到昌平国家实验室的支持和资助。
论文链接:
https://www.nature.com/articles/s41592-024-02239-0
参考文献:
1.Tan, L., Xing, D., Daley, N. & Xie, X.S. Three-dimensional genome structures of single sensory neurons in mouse visual and olfactory systems. Nature Structural & Molecular Biology 26, 297-307 (2019).
2.Kawaoka, J. & Lomvardas, S. LiMCA: Hi-C gets an RNA twist. Nature Methods (2024).
3.Wu, H. et al. Highly sensitive single-cell chromatin accessibility assay and transcriptome coassay with METATAC. Proceedings of the National Academy of Sciences 119, e2206450119 (2022).
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









好文章,谢谢分享。
14
#嗅觉受体# #三维基因组#
18