Nat Biotechnol:单细胞和空间转录组的高分辨率比对方法CytoSPACE,可重建复杂组织
2023-03-29 小鹿-先森 测序中国 发表于上海
CytoSPACE是一个通过全局优化来分析单细胞和空间转录组的工具。与其他方法不同,CytoSPACE确保全局最优的单细胞比对。
导语
单细胞的空间组织是细胞状态和行使功能的关键决定因素。例如,在人类肿瘤中,局部信号可以对单个细胞及其周围的微环境产生不同的影响,从而影响肿瘤的生长、进展和对治疗的反应。虽然空间转录组学(Spatial Transcriptomics, ST)已经成为描述原代组织标本中空间基因表达的强大工具。目前,已经开发了几种计算方法来推断给定ST样本中的细胞组成。这些方法大都使用来自单细胞转录组测序(scRNA-seq)数据作为参考,将ST的单位数据反卷积成细胞类型比例的矩阵。但这些方法仍缺乏单细胞分辨率,阻碍了空间定义的细胞状态、相互作用模式等生物学现象的发现。
为了应对这一挑战,近日,斯坦福大学的研究团队在Nature Biotechnology发表了题为“High-resolution alignment of single-cell and spatial transcriptomes with CytoSPACE”的文章。研究团队报道了CytoSPACE(Cellular Cyto Spatial Positioning Analysis via Constrained Expression Alignment),一种将单个细胞从单细胞转录组测序图谱映射到空间表达谱的优化方法。使用该方法得到的具有高基因覆盖率和空间分辨率的重建组织样本,还适用于下游分析,包括发现上下游依赖的细胞状态。此外,研究人员还在不同的平台和组织类型中,证实了CytoSPACE在噪声耐受性和准确性方面均优于已有的方法,并且能够在单细胞分辨率下进行空间组织分析。

文章发表在Nature Biotechnology
主要研究内容
CytoSPACE主要分为四个步骤。首先,为了解释scRNA-seq和ST数据在每种细胞类型的细胞数量上的差异,需要两个参数:ST样本中每种细胞类型的丰度分数和每个点的细胞数量。通过这两个参数,scRNA-seq数据集将被随机采样,以匹配ST数据集中每种细胞类型的预测细胞数。最后,CytoSPACE将每个细胞分配到空间坐标。
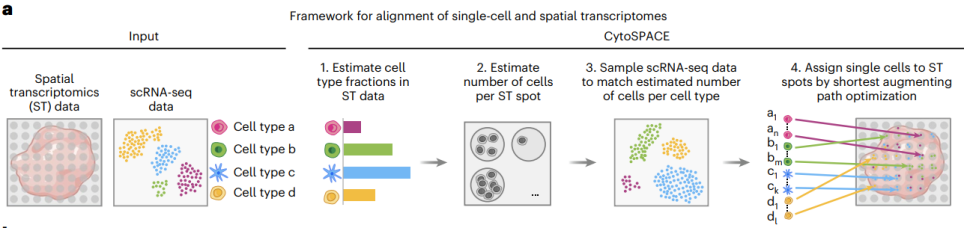
图1. CytoSPACE工作原理模式图,来源:Nature Biotechnology
研究团队利用模拟的单细胞ST数据集,对CytoSPACE与已有的其它12种方法进行了基准测试,包括Tangram和CellTrek。总的来说,在多个评估的噪声水平和细胞类型中,CytoSPACE在模拟ST数据集中将单个细胞映射到其已知位置时获得了比其他方法更高的精度。同时,CytoSPACE具有良好的稳健性。
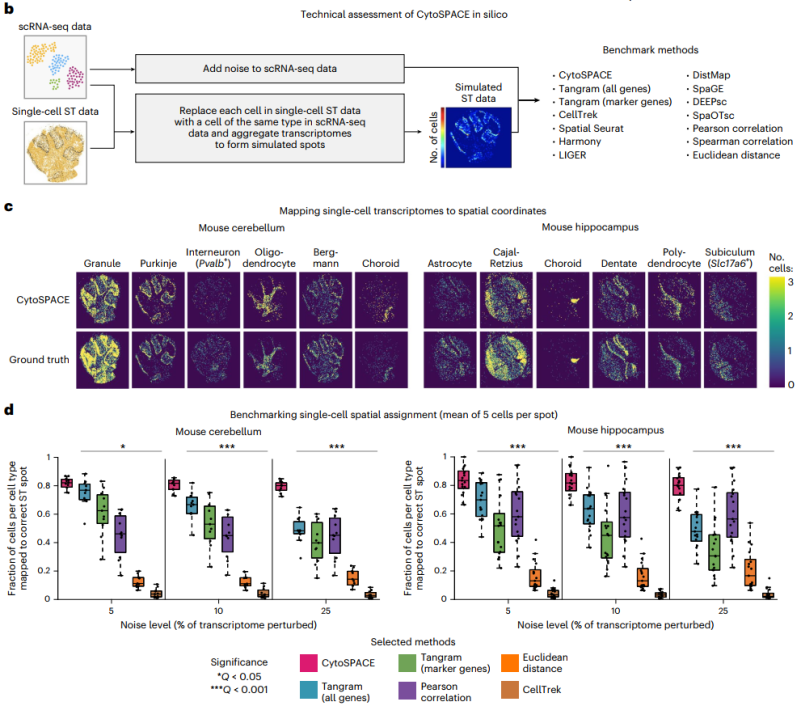
图2. CytoSPACE整体性能评估,来源:Nature Biotechnology
为了评估其应用于真实ST数据集的性能,研究团队检测了三种类型实体瘤(黑色素瘤,乳腺癌和结肠癌。)的原发肿瘤标本,包括6个ST样本数据和3个匹配肿瘤亚型的scRNA-seq数据集。结果显示,每个scRNA-seq数据集中的所有细胞类型都可被CytoSPACE比对上。此外,CytoSPACE非常高效,平均约5分钟即可处理一个Visum数据集。
在定量比较细胞状态的恢复与肿瘤微环境(TME)中的空间定位模式分析中,研究人员根据细胞类型中分配的细胞与肿瘤细胞的接近程度将其分为两组。分析结果与预期一致,在所有scRNA-seq和ST数据集组合中,CytoSPACE恢复了最接近癌细胞的CD4和CD8 T细胞中T细胞衰竭基因的空间富集。相比之下,Tangram和CellTrek生成的单细胞映射在预期方向上的T细胞衰竭基因富集程度要低得多,并且25%至33%的病例显示相反方向的富集。
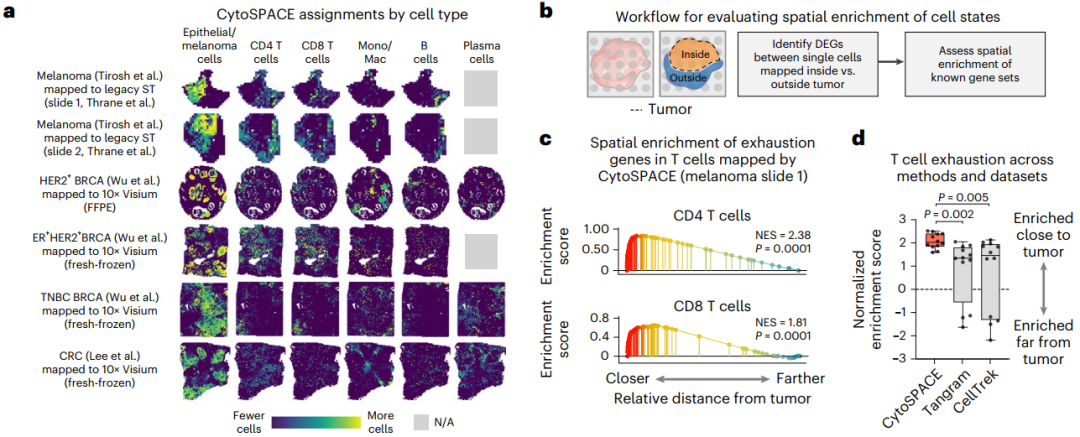
图3. CytoSPACE对空间定位模式的鉴定,来源:Nature Biotechnology
为了证明对其他空间偏向的细胞状态的适用性,研究人员将分析扩展到不同的TME谱系,发现细胞类型特异性基因的表达随着其与肿瘤细胞距离的变化而变化。同时,研究团队还评估了每种方法所映射的单细胞是否与CE9和CE10特异性的空间定位模式一致。结果发现,根据对T细胞衰竭因子的观察,CytoSPACE成功地恢复了淋巴和髓系CE9和CE10细胞的空间状态,在标记基因富集的大小和方向上都优于之前的12种方法。以上研究数据强调了CytoSPACE准确识别细胞状态的能力,包括那些仅从scRNA-seq或ST数据无法识别的状态。

图4. CytoSPACE准确识别细胞状态,来源:Nature Biotechnology
利用正常小鼠肾脏,在将包含30多个空间分辨(每个点直径55µm)的肾上皮亚型的scRNA-seq图谱映射到正常小鼠肾的10x Visium图谱后,研究人员评估了CytoSPACE是否再现了已知的空间组织模式。CytoSPACE重建了已知的带状区域并识别出优先共定位于肾小球的细胞类型,将近30个上皮状态排列在与其在肾单位上皮和集管系统中的已知位置一致的点上,并且优于以往的方法。
此外,研究团队还分析了一个乳腺癌样本,其中有超过55万个可注释的细胞和500个预先选择基因。分析证实,CytoSPACE可以准确地绘制所分析样本的单细胞,并概括了它们的空间依赖性。研究人员将一个scRNA-seq乳腺癌图谱映射到相同的数据集,除了观察到大多数标注的细胞类型具有很强的平台间一致性外,还证实了在肿瘤或邻近正常组织中富集的癌症相关T细胞具有显著偏差。总的来说,以上研究数据显示了CytoSPACE在单细胞水平上进行复杂组织重建的多功能性。
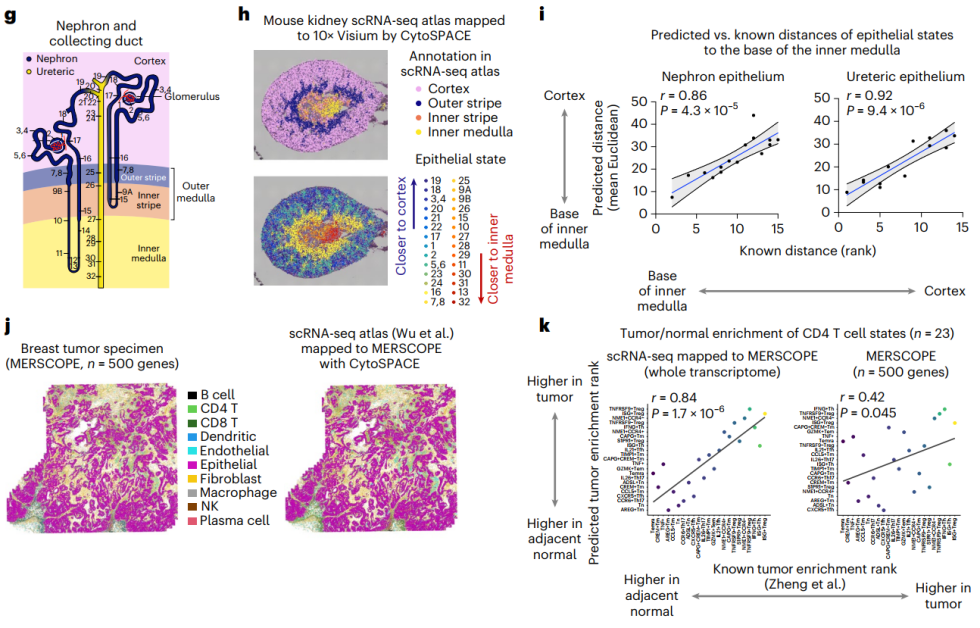
图5. CytoSPACE复杂组织的重建性能,来源:Nature Biotechnology
结语
综上所述,CytoSPACE是一个通过全局优化来分析单细胞和空间转录组的工具。与其他方法不同,CytoSPACE确保全局最优的单细胞比对。此外,在模拟数据集和真实数据集上,CytoSPACE的准确性明显高于已有方法。因此,预计CytoSPACE将被证明有助于在不同的生理和病理环境中破译单细胞空间变异和群落结构。
参考文献:
1. Vahid, M.R., Brown, E.L., Steen, C.B. et al. High-resolution alignment of single-cell and spatial transcriptomes with CytoSPACE. Nat Biotechnol (2023). https://www.nature.com/articles/s41587-023-01697-9
2. Cang, Z. & Nie, Q. Inferring spatial and signaling relationships between cells from single cell transcriptomic data. Nat. Commun. 11, 2084 (2020).
3. Hu, J. et al. Statistical and machine learning methods for spatially resolved transcriptomics with histology. Comput. Struct. Biotechnol. J. 19, 3829–3841 (2021).
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言