Nature:Illumina AI实验室等团队识别239种灵长类动物基因组的约束调控序列元件
2023-12-13 测序中国 测序中国 发表于上海
研究人员通过构建239种灵长类动物的全基因组多序列比对(MSA),分析了灵长类动物中约束序列元件(CSE)的特征。
非编码DNA是理解人类基因调控和复杂疾病的核心,分析进化约束序列可以了解人类基因组中调控元件的功能相关性。人类基因组与其他哺乳动物基因组之间的比较研究揭示了大量的约束基因和调控元件。但由于灵长类动物之间的进化距离很短,识别灵长类动物特有的受约束序列元件十分具有挑战性。目前,人类物种最终出现的系统发育分支所特有的选择性约束在很大程度上仍未确定。
近日,Illumina人工智能实验室联合西班牙庞培法布拉大学、贝勒医学院等机构的研究人员在Nature上发表了题为“Identification of constrained sequence elements across 239 primate genomes”的文章。研究人员通过构建239种灵长类动物的全基因组多序列比对(MSA),分析了灵长类动物中约束序列元件(CSE)的特征。利用这一资源,研究人员以5%的错误发现率,在灵长类动物和其他哺乳动物中确定了处于选择性约束下的人类调控元件,检测到111,318个DNase I超敏位点(DHS)和267,410个进化受限的转录因子结合位点,深度挖掘了人类等灵长类动物有别于其他哺乳动物的约束调控元件,这些调控元件富集了影响基因表达、复杂性状和疾病的人类基因突变。

文章发表于Nature
2023年6月,该研究团队发表在Science的文章中展示了233种灵长类动物的高覆盖率基因组数据,发现了错义突变的广泛存在。(点击查看此前报道)利用这些物种中大量良性错义突变的新目录,研究团队进一步开发并应用模型来识别蛋白质编码序列中的致病变异,这些变异仅占人类基因组的1%。在这项最新研究中,研究团队通过构建239个灵长类物种的全基因组多序列比对(MSA)来扩展先前的工作,以更好地表征人类基因组中的约束非编码调控序列。
Illumina人工智能副总裁Kyle Kai-How Farh表示:“我们发现了成千上万个在近期进化过程中才出现的调控元件,它们是灵长类动物和人类所特有的,在其他哺乳动物中不存在。”
239种灵长类动物全基因组比对
为了鉴定具有灵长类特异性约束的基因组元件,研究人员使用Megahit的测序数据组装了187个灵长类物种的基因组,通过与已发表的52个高质量灵长类动物参考基因组结合,建立了239种灵长类动物的无参考全基因组MSA,代表了所有主要的灵长类谱系,代表86%的属和所有16个科(图1a,b)。
研究证实,人类基因组的每个碱基平均被174个其他灵长类物种覆盖,且85%的常染色体区域被至少100个其他灵长目物种覆盖(图1c)。在考虑种内变异后,由装配和测序错误导致的平均剩余错配率降低到0.04%。研究人员将灵长类MSA与Zoonomica项目中其余的哺乳动物目相结合,生成了441种哺乳动物的MSA。这是迄今为止全基因组MSA中对哺乳动物最深入的物种采样,并且可以在广泛的哺乳动物和灵长类动物中检测序列约束性。
灵长类动物的约束蛋白质编码序列
将MSA中可用的灵长类物种数量增加到239种,使系统发育分支长度比之前Zoonomia研究中可用的43种灵长类物种增加2.8倍。研究人员使用phyloP评估了MSA区域全基因组的碱基约束性,发现人类基因组中3.1%的碱基在所有灵长类动物中受到约束,而在相同阈值下,240种哺乳动物中有7.1%的碱基受到约束。
同时,研究团队使用phasstcon在灵长类动物中检测了157Mb的约束序列元件,发现蛋白质编码DNA(包括外显子、启动密码子和终止密码子)在phascons元件中富集程度很高;包含转录区和可及染色质的顺式调控元件(CRE)或转录因子占据的非编码DNA也显著富集。密码子约束具有的周期性模式,该模式在核苷酸水平上可以区分外显子与附近的内含子序列(图1e)。研究人员在灵长类动物中鉴定出有约束证据的179,329个外显子,其中99%的外显子在非灵长类哺乳动物和脊椎动物中广泛约束,在灵长类动物中有2178个特异性约束外显子。大多数灵长类动物的约束外显子(72%)在小鼠基因组的同源区域被注释为蛋白质编码,表明它们不是新进化的编码序列,而是在灵长类动物的选择性约束中发生了进化。以上结果表明,从现有序列进化出新的蛋白质编码基因或外显子是罕见的,而先前存在的外显子的功能重要性增加是一种相对常见但仍然罕见的事件。
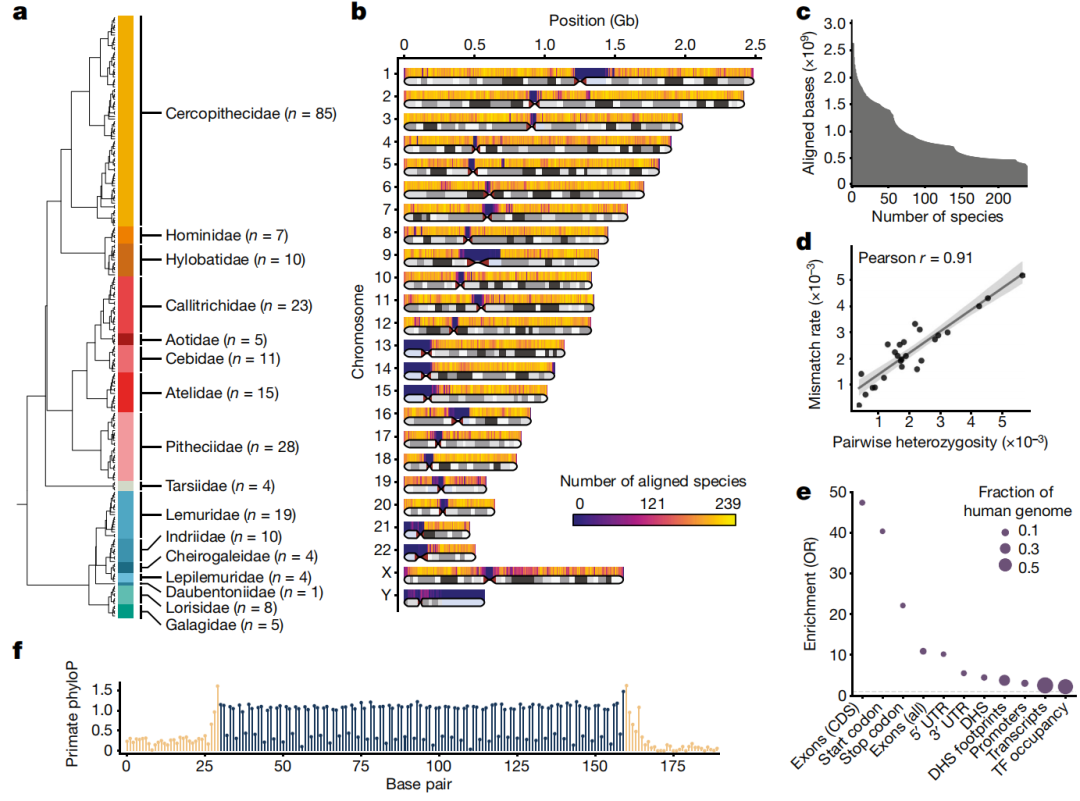
图1.239种灵长类动物的MSA,来源:Nature
灵长类动物的约束顺式调控元件(CRE)
研究人员在438种细胞类型的120万个DNA酶I超敏位点(DHS)元素的高分辨率图谱中,估计了灵长类和哺乳动物的平均序列约束性。结果发现,在1亿年前分化的物种中,42%的物种存在序列约束证据,11%在灵长类动物中存在显著约束证据,但在哺乳动物或脊椎动物中缺乏约束证据(图2a,b)。
在这些DHS元件中,转录因子的占据阻止了DNA酶I切割,从而在核苷酸分辨率上产生转录因子结合或转录因子结合事件(TFBS)。在360万个TFBS足迹中,30%具有哺乳动物广泛约束证据,8%显示灵长类特异性约束。值得注意的是,66%灵长类动物特异性约束的DHS元件具有在哺乳动物中保守的TFBS,表明调控功能最初是在共同祖先中进化的(图2c)。
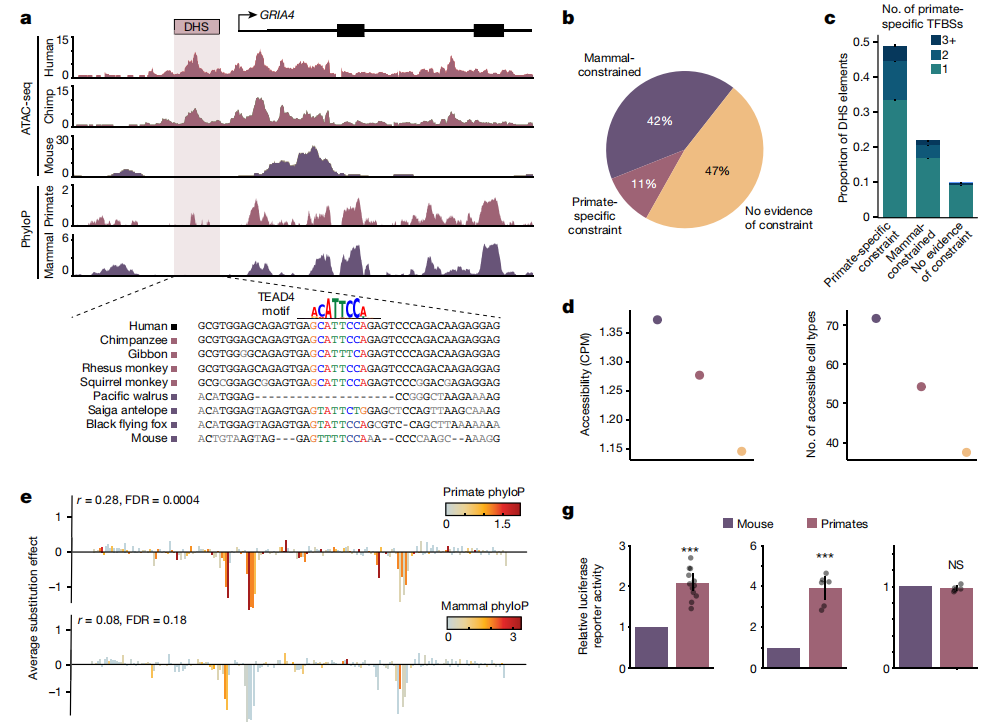
图2.具有灵长类动物特异性约束的非编码调控元件的鉴定,来源:Nature
接下来,研究人员探索了现代人类群体中破坏灵长类约束调控元件的基因突变的选择证据。结果发现,预测的灵长类动物特异性元件靶基因的功能缺失突变明显少于预期(图3a)。此外,在非编码灵长类特异性约束元件中观察到突变约束增加(图3b),表明灵长类动物中特有的约束调控元件在人类中具有重要的顺式调控功能。
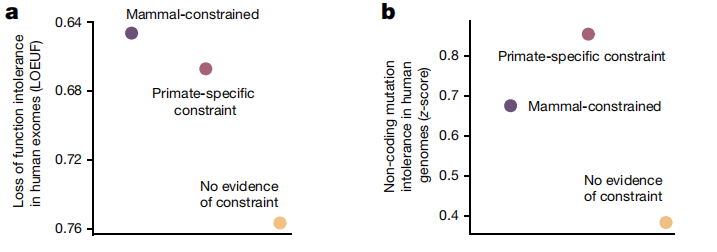
图3.约束调控元件的特征,来源:Nature
研究人员还在人类基因组中发现了7460万个在239种灵长类动物中完全约束的位点。进一步分析发现,临床表型和复杂性状的精细定位突变在所有类别的远端可及染色质元件和足迹中都得到了富集,包括具有灵长类特异性约束的突变(图4a)。影响高度约束基因表达的变异倾向于在更深度约束的DHS元件和足迹,而影响约束较少的基因表达的突变往往驻留在具有较新约束的元件上(图4b)。CRE中12%的精细映射突变仅在灵长类动物中受到约束,而在胎盘哺乳动物中没有受到约束,有93个可能致病的调控突变与人类复杂性状和临床表型相关。
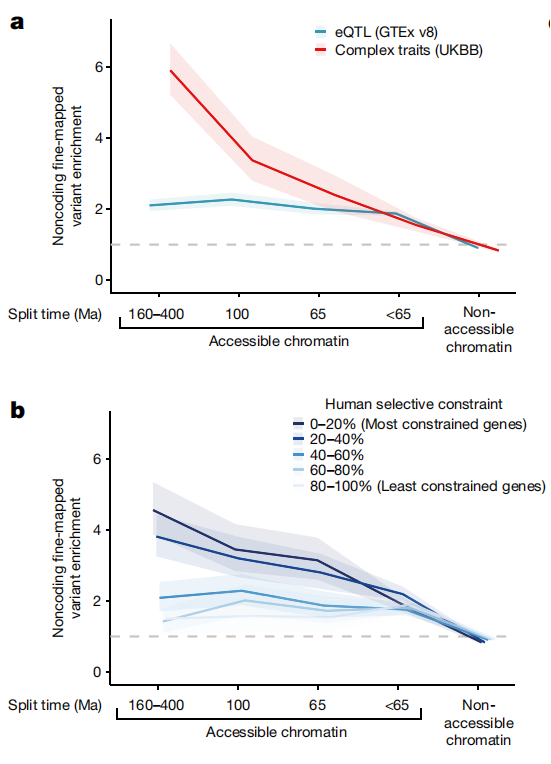
图4.在受约束的非编码CRE中富集复杂性状变异,来源:Nature
研究团队通过将239种灵长类动物的基因组(包括187种新组装的灵长类动物),鉴定了数十万个约束非编码序列元件。这些CRE是独特的进化记录,提供了一个视角来观察导致物种近期进化的机制。研究发现,许多以前没有显示序列约束证据的人类CRE实际上只在灵长类动物中受到限制,这大大扩展了人类基因组中已知的约束非编码元件的数量。
灵长类动物中的序列约束独特地预测了调节元件子集的功能,并且特定约束元件在不同人类细胞类型和不同灵长类动物物种中具有更高和更相似的调节功能。“该研究加深了我们对非编码基因组中疾病相关变异的了解,”Farh表示。“非编码DNA比蛋白编码序列进化得更快,我们发现人类常见疾病背后的遗传变异也往往集中在这些新进化的调控序列上。”
根据该研究结果,Illumina计划通过Illumina Dragen、Emedgene、Connected Insights和Illumina Connected Analytics工具向客户提供全基因组约束评分。此外,随着未来对更多灵长类动物的测序、组装和分析,约束序列元件数据库有望进一步完善。
论文原文:
Kuderna, L.F.K., Ulirsch, J.C., Rashid, S. et al. Identification of constrained sequence elements across 239 primate genomes. Nature (2023). https://doi.org/10.1038/s41586-023-06798-8
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言











#非编码DNA# #全基因组多序列比对# #约束序列元件#
57