四川大学华西药学院王乾韬小组JCIM论文:微秒级分子动力学模拟驱动MTDH-SND1蛋白-蛋白相互作用抑制剂的发现
2023-10-16 ComputArt计算有乐趣 ComputArt计算有乐趣 发表于上海
蛋白-蛋白相互作用是调控生物过程的重要媒介,与癌症等多种疾病密切相关。但PPI作用界面往往具有动态、宽泛和缺乏明确结合口袋等特点,导致PPI抑制剂的合理药物设计具有较大挑战。
蛋白-蛋白相互作用(protein-protein interactions,PPIs)是调控生物过程的重要媒介,与癌症等多种疾病密切相关。但PPI作用界面往往具有动态、宽泛和缺乏明确结合口袋等特点,导致PPI抑制剂的合理药物设计具有较大挑战。
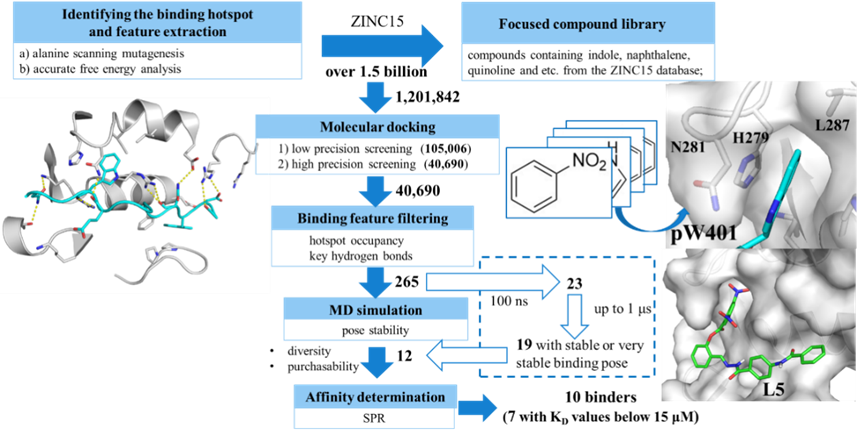
针对这一问题,该研究以MTDH-SND1蛋白-蛋白相互作用为例,尝试探索了分子动力学(molecular dynamics, MD)模拟驱动的大规模虚拟筛选和实验验证,从12个测试化合物中成功得到了10个SND1蛋白的可结合化合物(binder),并初步验证了其中亲和力较高的L5对MTDH-SND1相互作用的阻断效果和体外抗肿瘤活性。结果显示L5可能是该靶点目前体外活性最好的小分子化合物,为后续研究提供了基础。相关研究成果近期发表于美国化学会出版的计算化学和化学信息学权威期刊Journal of Chemical Information and Modeling(J. Chem. Inf. Model. 2023, 63, 11, 3614–3627)。论文第一作者为四川大学华西药学院硕士徐亚迪,通讯作者为王乾韬副教授。
截止该文发表前,已报道的MTDH-SND1抑制剂只有一类小分子抑制剂(Nature Cancer, 2022, 3, 43)和三种多肽抑制剂(J Med Chem, 2022, 65, 12188; Molecular Cancer Therapeutics, 2021, 20, 76)(图1)。为找到亲和力更高的小分子抑制剂,怎样发挥微秒级MD模拟在探索结构动态上的优势,将其有效地融入虚拟筛选,提高PPI抑制剂的筛选效率和效果是该研究拟回答的核心问题。论文主要围绕以下方面展开。
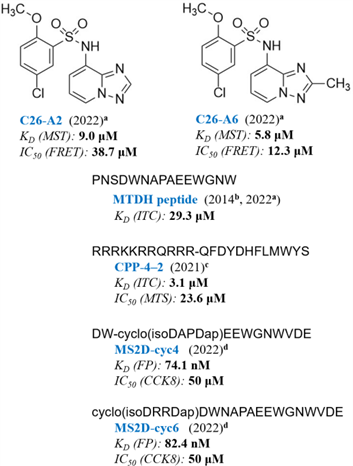
图1. 已知MTDH-SND1小分子和多肽抑制剂的结构和报道的亲和力
(一)自由能计算对氨基酸残基能量贡献定性/定量分析上的有效性
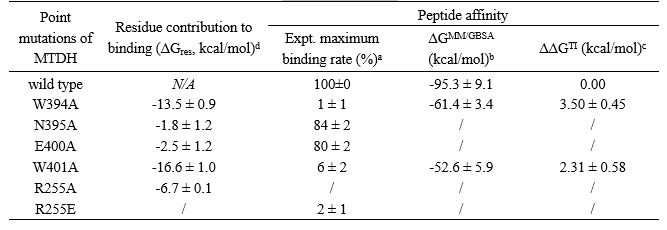
文献已知实验研究表明MTDH-SND1界面存在若干关键残基,包括W394、W401、R255等。以此为基础,作者首先考察了虚拟丙氨酸扫描、MM/GBSA和TI自由能计算方法在预测蛋白表面不同氨基酸残基对PPI的贡献,验证了三种方法在定性或定量分析方面的有效性,为后续研究提供了理论基础。
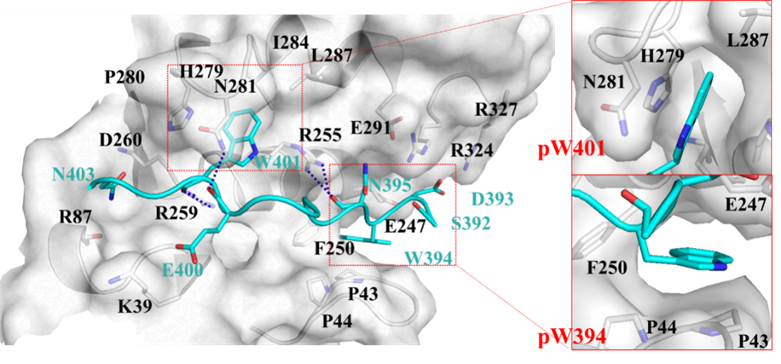
图2. MTDH肽(青色)和SND1蛋白(银色)的结合方式
表1. 不同氨基酸残基对MTDH多肽与SND1蛋白亲和力贡献的计算与文献实验结果对比
(二)微秒级MD模拟揭示热点口袋动力学特征,成为虚拟筛选关键决策条件
已知晶体结构显示,有MTDH多肽结合的SND1表面的两个口袋(pW401和pW394)体积有限(~250 Å3)且相距较远(~15 Å)。因此,在进行药物筛选和设计之前,必须在这两个口袋之间做出选择。
通过SND1蛋白在有无配体情况下的多次1 μs普通MD和GaMD模拟, 发现pW401口袋体积在无配体情况下缩小约一半到100 Å3左右,而pW394口袋则缩小到几乎消失。提示pW394可能比pW401更为“隐蔽”,配体将其打开(或“遇到”其打开状态)的可能性更低。基于此考虑,即使已有的实验结果和作者的TI自由能计算结果均提示占据pW394口袋的色氨酸残基对PPI亲和力贡献更大,作者仍然认为pW401口袋可能是更可能的小分子结合位点,并以此为基础开展了后续虚拟筛选。随后2022年于Nature Cancer报道的C26-A6化合物与SND1的晶体结构也从侧面证明了该假设的正确性。
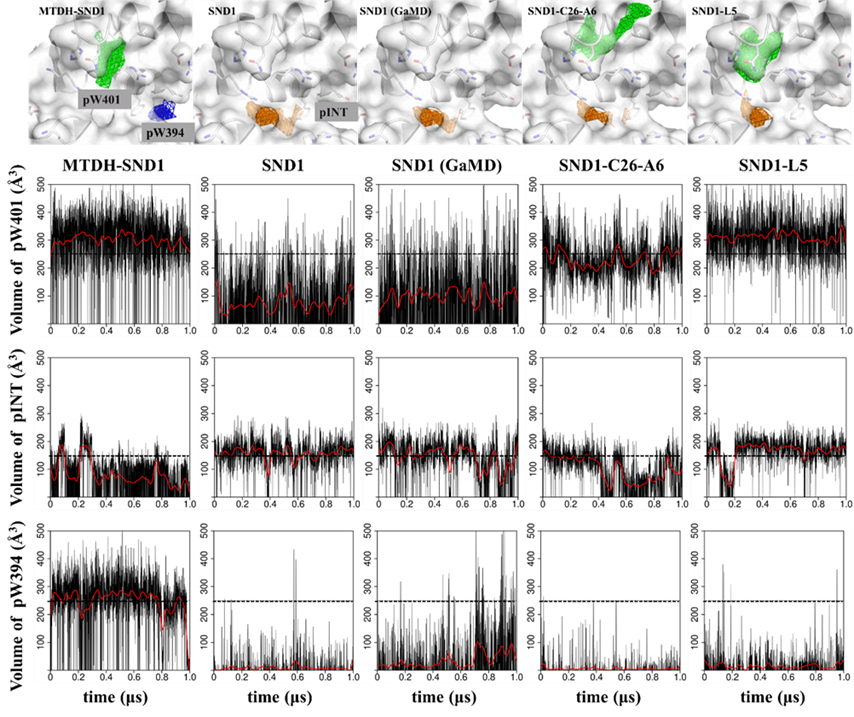
图3. SND1蛋白表面口袋在1 μs MD和GaMD中的体积变化情况
(三)筛选流程
基于色氨酸对结合具有关键作用的假设,作者对ZINC15约15亿化合物进行初筛得到一个约120万具有类似结构特征(吲哚、喹啉、萘、氯苯、硝基苯等)的焦点化合物库进行后续对接筛选。
分子对接使用AutoDock Vina进行两轮对接,得到约4万个化合物。同时,通过对少数对接分子进行初步MD模拟,研究其结合特征,作者选择了两个较为共性的特征用于对对接结果的再筛选:分别为疏水弹头是否占据pW401口袋以及是否能够与R255等热点残基形成氢键,进一步将目标分子减少到约265个。最后,进行100 ns 到1μs的MD模拟以评估结合姿势的稳定性。其中19个化合物被认为具有稳定或较为稳定的结合姿势。
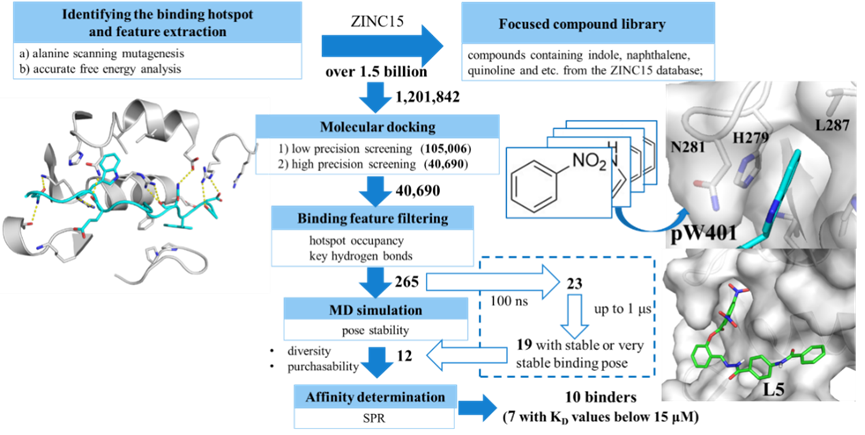
图4. 筛选流程示意图
(四)实验验证
基于化学结构多样性和化合物可购买性,从19个化合物选择购买了12个(L1-L12)进行SPR亲和力测试。并以L5为例,进一步预测了其与SND1可能的结合模式;在细胞水平验证其抗肿瘤活性及其对MTDH-SND1的阻断作用。
综上所述,作为虚拟筛选得到的迄今为止与SND1亲和力最高的已知小分子,L5的报道表明MD驱动的筛选策略可能是一个有效的 PPI抑制剂筛选方法,有望进一步促进MTDH-SND1抑制剂的探索。但现有结果并不能排除L5脱靶的可能性,有必要进一步探索其作用机制。
表2. SPR亲和力结果
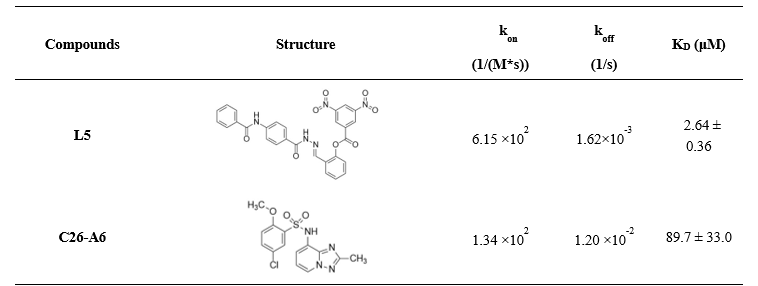
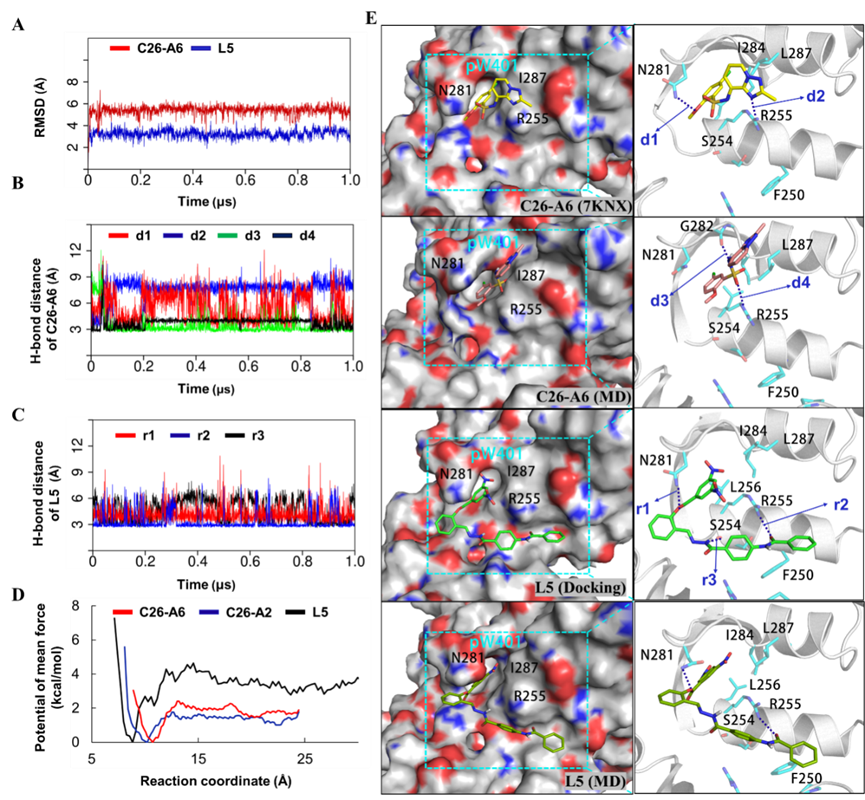
图5. L5和C26-A6与SND1蛋白结合构象比较及伞形采样PMF结果
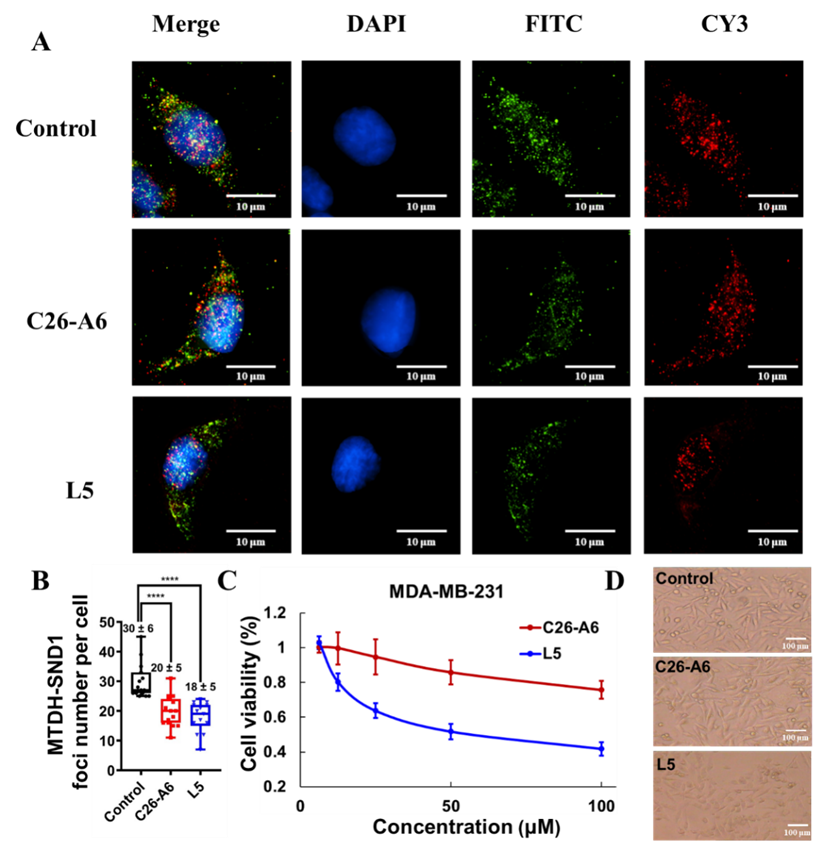
图6. L5阻断MTDH-SND1相互作用,抑制MDA-MB-231乳腺癌细胞的增殖
部分参考文献
Shen, M. et al, Small-molecule inhibitors that disrupt the MTDH-SND1 complex suppress breast cancer progression and metastasis. Nature Cancer 2022, 3 (1), 43-59.
Chen, H. et al Structure-Based Design, Optimization, and Evaluation of Potent Stabilized Inhibitors MTDH and SND1 Interaction. Journal of Medicinal Chemistry 2022, 65 (18), 12188-12199.
Li, P. et al, Disruption of SND1-MTDH Interaction by a High Affinity Peptide Results in SND1 Degradation and Cytotoxicity to Breast Cancer Cells In Vitro and In Vivo. Molecular Cancer Therapeutics 2021, 20 (1), 76-84.
Guo, F. et al, Structural Insights into the Tumor-Promoting Function of the MTDH-SND1 Complex. Cell Reports 2014, 8 (6), 1704-1713.
Wan, L. et al, MTDH-SND1 Interaction Is Crucial for Expansion and Activity of Tumor-Initiating Cells in Diverse Oncogene- and Carcinogen-Induced Mammary Tumors. Cancer Cell 2014, 26 (1), 92-105.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言







