Nature Structural & Molecular Biology:施一公团队揭示酵母组蛋白去乙酰酶复合体Rpd3L的组装机制
2023-04-21 “生物世界”公众号 “生物世界”公众号 发表于上海
该论文报道了酿酒酵母组蛋白去乙酰酶复合体Rpd3L的结构,揭示了其组装机制,提出了其靶向染色质的工作模型,并为设计高特异性去乙酰酶抑制剂提供坚实的分子基础。
组蛋白乙酰化在调节基因表达和染色质结构方面起着重要作用。细胞核中组蛋白的乙酰化水平由乙酰转移酶(histone acetyltransferase,HAT)和去乙酰化酶(histone deacetylase,HDAC)共同调控。HDAC抑制剂已被用于多种疾病或癌症的临床治疗。然而,由于HDAC活性位点的结构相似性,目前上市的HDAC抑制剂的特异性有限。研究发现锌离子依赖的HDAC一般作为催化亚基存在于多种转录调控复合体中,如SIN3-HDAC、NuRD、CoREST、SMRT/NCoR及MiDAC等。人们渐渐意识到这些复合体对于将HDAC定位于特异染色质区域并实现时空的精准调控至关重要。
SIN3-HDAC复合体作为其中重要的一员,从酵母到人源都是保守的,参与了许多细胞过程,包括发育、生长、分化和衰老等等此外,它们还为多种癌症提供了重要的治疗靶标,如三阴性乳腺癌和胰腺癌等。人源Sin3蛋白的杂合突变或缺失可能导致伴有颅面缺陷的智力障碍综合症。由于缺乏关键的结构信息,长期以来人们对SIN3-HDAC复合体的理解较为滞后。
2023年4月20日,西湖大学生命科学学院施一公团队在 Nature 子刊 Nature Structural & Molecular Biology 上发表了题为:Structure of a SIN3-HDAC complex from budding yeast 的最新研究论文。
该论文报道了酿酒酵母组蛋白去乙酰酶复合体Rpd3L的结构,揭示了其组装机制,提出了其靶向染色质的工作模型,并为设计高特异性去乙酰酶抑制剂提供坚实的分子基础。


研究团队从酿酒酵母中提取了内源的Rpd3L复合体,并利用单颗粒冷冻电镜技术解析了该复合体的结构,平均分辨率为 2.6 ?(图1)。结合高质量冷冻电镜密度图与交联质谱结果,研究人员搭建了含有12个亚基的结构模型(其中包括各两分子的Sin3和催化亚基Rpd3)。结构分析发现整个复合体由"T "形支架连接ARM1和ARM2形成。
在每个ARM中,Sin3与不同的亚单位相互作用,为组蛋白去乙酰化酶Rpd3创造不同的 “环境”:ARM1处于抑制状态,Rpd3的酶活中心被封锁(图2);而ARM2处于开放构象,Rpd3’的酶活中心暴露在外。结合这些结构特征和以往的报道,研究人员提出了Rpd3L复合体靶向特异染色质区域的工作模型。
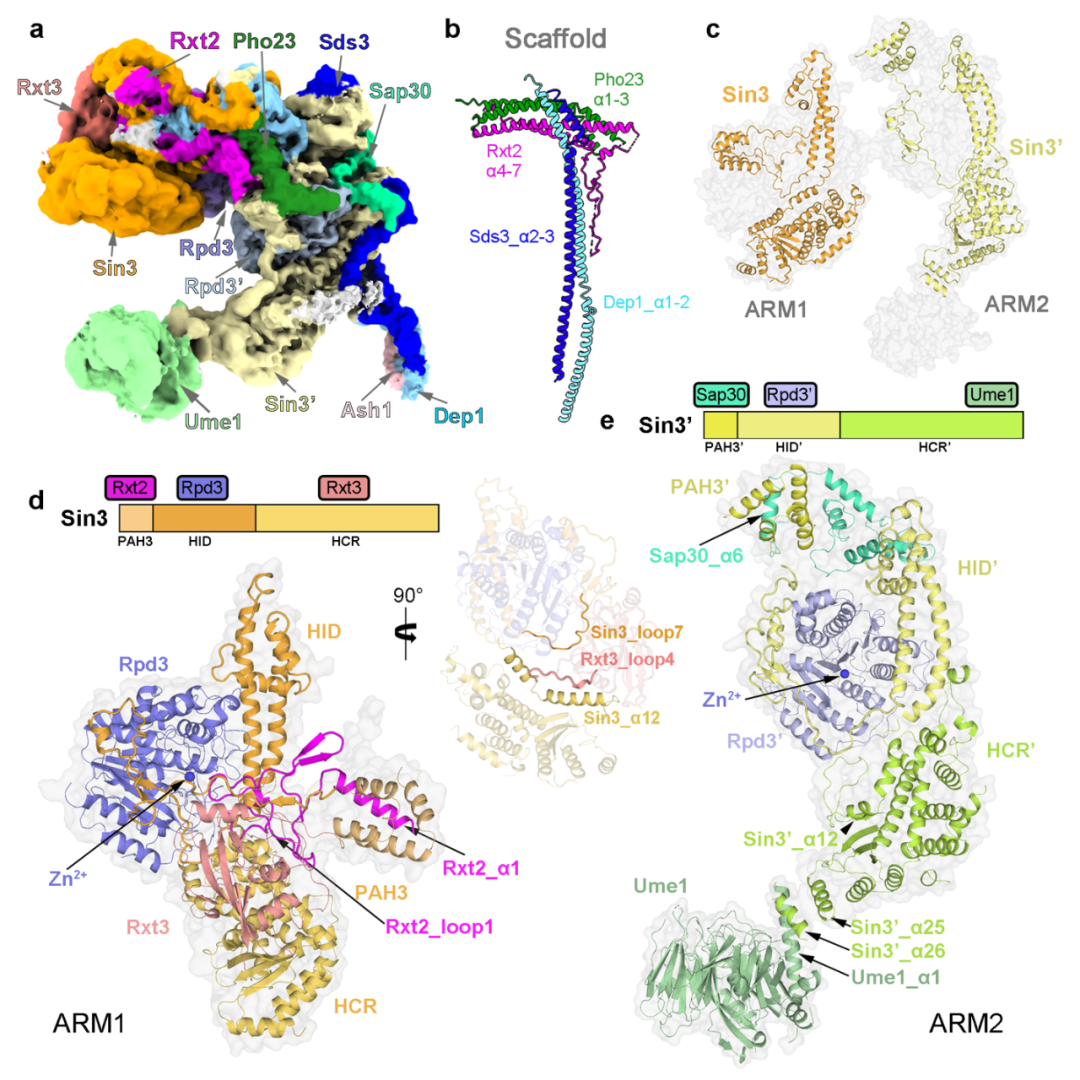
图1 酿酒酵母Rpd3L的结构

图2 ARM1中Rpd3的酶活中心被Rxt2的第80位亮氨酸残基占据
另外,Rpd3L复合体的不对称结构与其他已发表去乙酰酶复合体的结构有很大的差异。许多基于细胞和动物的研究表明不同的HDAC复合体具有不同的生物活性,影响不同的生理和发育功能。这意味着以HDAC复合体为目标设计药物可能获得高特异性。然而,人源SIN3-HDAC复合体的现有结构信息非常有限。酵母Rpd3L复合体的结构解析代表着对SIN3-HDAC复合体的理解向前迈出了一大步,并为设计高特异性药物提供潜在的策略。
西湖大学生命科学学院施一公团队副研究员王程程、占谢超为论文共同通讯作者,西湖大学博士生郭舟妍和储晨为论文共同第一作者。施一公教授为该研究提供了大力支持和建议。西湖大学博士生卢怡辰、原西湖大学助理研究员张晓峰、西湖大学理学院研究员吴明轩和博士生肖义杭参与了本研究的部分工作。北京协和医院黄超兰教授和高帅鑫高级工程师参与了质谱部分的工作。
原始出处:
Guo, Z., Chu, C., Lu, Y. et al. Structure of a SIN3–HDAC complex from budding yeast. Nat Struct Mol Biol (2023). https://doi.org/10.1038/s41594-023-00975-z.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








很受启发
75