Nat Commun|俞云松/陈衍课题组发文建立追踪头状葡萄球菌多重耐药菌新方法
2022-07-29 浙江大学医学院 网络
在本研究中课题组开发并建立了头状葡萄球菌cgMLST系统,揭示了利奈唑胺耐药头状葡萄球菌的克隆传播和耐药机制。
2022年7月22日,浙江大学医学院附属邵逸夫医院感染科俞云松/陈衍课题组在《Nature Communications》发表题为Development of a novel core genome MLST scheme for tracing multidrug resistant Staphylococcus capitis的研究论文。该研究首次建立了头状葡萄球菌核心基因组多位点分型(cgMLST)系统,并且利用此分子分型流程发现,利奈唑胺耐药头状葡萄球菌已经在临床广泛传播,产生重要威胁。

头状葡萄球菌(Staphylococcus capitis)是一种常见的凝固酶阴性葡萄球菌,因常分离于人类的头、面、颈部而得名。该菌作为一种条件致病菌可导致各种严重感染,如乳腺炎、复杂皮肤软组织感染、人工关节感染、心内膜炎、起搏器相关感染、新生儿医院获得性迟发性脓毒症等。
核心基因组多位点分型系统(cgMLST)是微生物领域常用的分型系统,具有分辨率高、操作简单、运行效率高、标准化等优点。既往的cgMLST建立策略存在纳入核心基因数量不多、核心基因准确率低等问题。针对这些问题,课题组使用了泛基因组分析软件Panaroo,并应用了3个不同的基因组集:初始基因组集用于构建初始核心基因组,验证基因组集用于筛选核心基因,检验基因组集用于检验cgMLST系统。课题组从公共基因组数据库中收集头状葡萄球菌基因组,经过质控后作为初始基因组集。对初始基因组集进行核心基因组分析,确定初始核心基因组,接着进行初步筛选和验证基因组集筛选后最终建立包含1492个基因的cgMLST系统。
为了验证该cgMLST系统,课题组收集所有利奈唑胺耐药头状葡萄球菌的基因组作为检验基因组集,纳入菌株包括分离自杭州、上海和哈尔滨的菌株。检验基因组集与验证基因组集共同进行cgMLST分析(图一)和基于核心基因组SNP的系统发育分析(图二)。
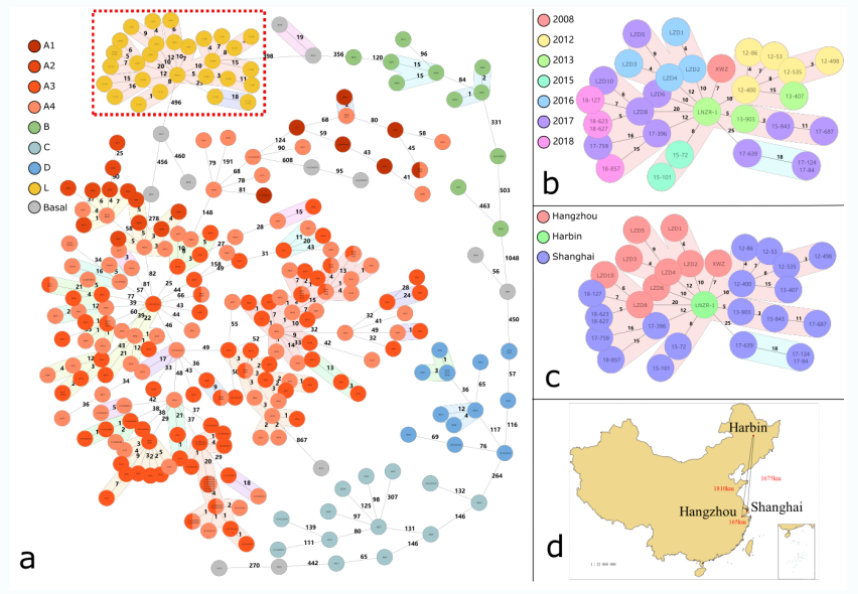 图一 验证基因组集和检验基因组集的cgMLST分析结果
图一 验证基因组集和检验基因组集的cgMLST分析结果
尽管菌株之间时空差异很大,绝大多数利奈唑胺耐药头状葡萄球菌均属于同一克隆,我们将这一克隆命名为L克隆。其他菌株在cgMLST分析结果中可以大致区分为4个主要克隆,将其命名为克隆A、克隆B、克隆C和克隆D。(图一)

图二 验证基因组集和检验基因组集的系统发育树及耐药基因分布情况
结果证实,基于核心基因组SNP的系统发育分析的区分结果与cgMLST结果一致。(图二)大部分L克隆菌株携带cfr耐药基因(介导利奈唑胺耐药),该基因由质粒pLZD8_2携带,并且只存在于L克隆中。另外,这些菌株中23S rRNA多拷贝突变(G2576T、C2104T)也是利奈唑胺耐药机制之一。(图二)
利奈唑胺耐药的头状葡萄球菌在我国已有不少报道,但缺乏多中心菌株的亲缘关系分析研究。我们的cgMLST分析鉴定发现了重要耐药克隆L克隆。虽然L克隆目前只发现于中国,但在One health背景下,多重耐药菌跨种属、跨国境传播已经成为全球性的危机,亟需进行更大范围的国际间的流行病学调查来监测L克隆的全球性流行和传播。在完善的cgMLST系统的帮助下,我们可以更好地监测多重耐药头状葡萄球菌克隆播散情况。
综上,在本研究中课题组开发并建立了头状葡萄球菌cgMLST系统,揭示了利奈唑胺耐药头状葡萄球菌的克隆传播和耐药机制,为多重耐药病原体的流行病学监测提供了可靠的分子分型工具。
浙江大学医学院感染病学博士研究生王桢干、顾超(嘉兴市第一医院呼吸科)和浙江大学医学院附属邵逸夫医院感染科孙璐博士为本文的共同第一作者,浙江大学医学院附属邵逸夫医院俞云松教授、陈衍副主任医师为本文的共同通讯作者。该课题的开展依托于浙江省微生物技术与生物信息研究重点实验室等研究平台。
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#新方法#
69
#Nat#
0
#COMMUN#
56
#耐药菌#
55
#多重耐药#
103
#葡萄球菌#
51
#多重耐药菌#
0