Cell封面:David Liu再取新突破:不需要做实验,就能知道基因编辑的结果
2020-07-28 iNature iNature
尽管碱基编辑器被广泛用于目标点突变,但是决定碱基编辑结果的因素尚不十分清楚。
尽管碱基编辑器被广泛用于目标点突变,但是决定碱基编辑结果的因素尚不十分清楚。
2020年7月23日,博德研究所David R. Liu团队在Cell 在线发表题为“Determinants of Base Editing Outcomes from Target Library Analysis and Machine Learning”的研究论文,该研究在哺乳动物细胞中38,538个基因组整合靶标上表征了11个胞嘧啶和腺嘌呤碱基编辑器(CBE和ABE)的序列-活性关系,并使用所得结果训练了BE-Hive,这是一种机器学习模型,可准确预测碱基编辑基因型结果(R ≈0.9)和效率(R≈0.7)。
研究人员以≥90%的准确度纠正了3388个与疾病相关的SNV,其中包括675个等位基因,其“旁观者”核苷酸被BE-Hive正确预测,因此无法编辑。该研究发现了以前无法预测的C-to-G或C-to-A编辑的决定因素,并利用这些发现以≥90%的准确性纠正了174个病原性SNV的编码序列。最后,该研究利用BE-Hive的见识来设计新颖的CBE变体,以调节编辑结果。这些发现启发了碱基编辑,实现了以前难以处理的目标的编辑,并为新的基础编辑器提供了改进的编辑功能。
另外,2020年7月8日,博德研究所David R. Liu及华盛顿大学医学院Joseph D. Mougous共同通讯在Nature 在线发表题为“A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing”的研究论文,该研究描述了一种细菌间毒素,将其命名为DddA,它可以催化dsDNA中胞苷的脱氨。该研究设计了无毒且无活性的split-DddA半分子:DddA分割的一部分结构域(转录激活子样效应子阵列蛋白)和尿嘧啶糖基化酶抑制剂的融合,产生了无RNA的DddA衍生的胞嘧啶碱基编辑器(DdCBE),可催化人mtDNA中的C?G到T?A转化,具有高靶标特异性和产品纯度。该研究使用DdCBEs建模人类细胞中与疾病相关的mtDNA突变,从而导致呼吸速率和氧化磷酸化的改变。不含CRISPR的DdCBE可以精确操纵mtDNA,而不是消除因被靶向核酸酶切割而产生的mtDNA拷贝,这对线粒体疾病的研究和潜在治疗具有广泛的意义。
2020年6月29日,博德研究所David Liu在Nature Biotechnology 在线发表题为“Programmable m6A modification of cellular RNAs with a Cas13-directed methyltransferase”的研究论文,该研究证明了具有截短的METTL3甲基转移酶结构域或者是METTL3:METTL14甲基转移酶复合物与核定位dCas13融合体,可以对细胞质RNA进行特异性m6A掺入,而前者的融合蛋白脱靶活性特别低。跨多个位点的独立细胞测定法证实,这种靶向RNA甲基化(TRM)系统以高特异性介导了有效的m6A安装在内源RNA转录物中。最后,该研究表明TRM可以诱导m6A介导的转录本丰度变化和选择性剪接。这些发现将TRM确立为用于靶向转录组工程的工具,可以揭示单个m6A修饰的作用并分析其功能作用。
2020年6月22日,博德研究所David Liu团队在Nature Biotechnology 在线发表题为“Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors”的综述文章,该综述首先描述已表征的Cas9和Cas12核酸酶的天然变异体,并详细介绍具有扩大的靶向范围和特异性的Cas9和Cas12核酸酶变异体的开发。接下来,该综述讨论碱基编辑器的开发和应用,这些编辑器可精确安装点突变而无需双链DNA断裂(DSB)或供体DNA模板。最后,该综述总结了新兴的CRISPR–Cas基因组编辑工具,包括介导大片段DNA重排的Cas转座子和重组酶,以及主要编辑器,它们以取代原始DNA序列的方式直接将编辑后的序列复制到目标DNA位点。
基因组DNA中靶向核苷酸的编辑是研究和治疗应用的一项关键功能。单核苷酸变体(SNV)约占已知致病等位基因的一半,因此有针对性的点突变可以促进遗传病的研究或潜在治疗。以前,研究人员开发了胞嘧啶碱基编辑器(CBE)和腺嘌呤碱基编辑器(ABE),它们共同实现了所有四个过渡点突变的靶向(C→T,T→C,A→G ,以及G→A),并具有较高的期望取代率与不期望的插入和缺失(indels)比率。
碱基编辑的实用性启发了具有不同属性的碱基编辑器变体的开发。迄今为止,通过分析少量基因组位点的编辑结果来收集这些属性,通常选择这些位点与先前的基因组编辑研究相一致。但是,碱基编辑器和目标序列之间的相互作用会以复杂的,有时是不直观的方式影响编辑结果。结果,获得具有所需效率的所需基因型通常需要对每个靶标进行碱基编辑和单向导RNA(sgRNA)选择的经验优化。
某些不适合用于碱基编辑的规范准则的可行目标可能会被忽略,因为用于目标选择的简单准则无法完全捕获碱基编辑的范围。对碱基编辑的序列和脱氨酶决定因素进行系统,全面的分析将增强我们对碱基编辑器的理解,促进它们在精确编辑应用程序中的使用,并指导新的碱基编辑器的开发。
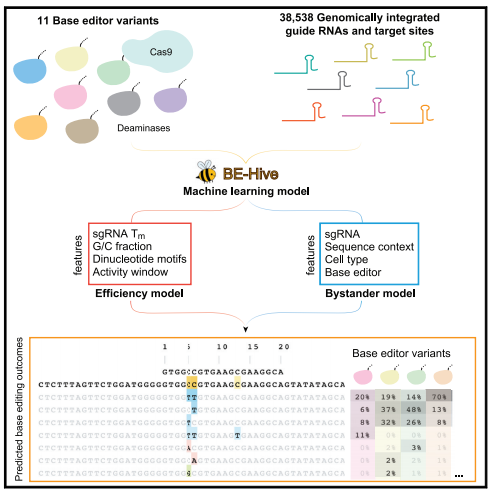
文章模式图(图源自Cell )
在这项研究中,研究人员开发了包含38,538对sgRNA和靶序列对的文库,并将它们整合到三种哺乳动物细胞类型的基因组中,以全面表征8种流行CBE和ABE的碱基编辑结果和序列-活性关系。研究人员分析了脱氨酶,序列背景和细胞类型在确定碱基编辑产生的基因型中的作用,并开发了一种机器学习模型,可以在任何目标位置准确预测碱基编辑结果,包括许多以前不可预测的特征。
利用所得信息,研究人员应用了各种碱基编辑器(包括新近设计的变体),将3388个与疾病相关的SNV的基因型和2399个编码序列准确地校正为野生型(≥90%的精度),包括通过非规范的碱基编辑结果。这些发现大大扩展了我们对碱基编辑的理解,并揭示了新的和先前描述的碱基编辑器的新功能。
原始出处:
Andrew V. Anzalone, Luke W. Koblan & David R. Liu, et.al. Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors. Nature Biotechnology volume 38, pages824–844(2020)
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言













#CEL#
42
#Cell#
54
#基因编辑#又有突破
158
#新突破#
48