Nature:检测血液中微生物DNA,癌症早诊断
2020-03-15 顾露露 生物探索
当Gregory Poore还是一名大一新生时,他原本健康的祖母被诊断患有晚期胰腺癌,12月下旬确诊,次年1月就去世了。这让他感到震惊,“因为她几乎没有任何征兆或症状,为什么她的癌症没有更
当Gregory Poore还是一名大一新生时,他原本健康的祖母被诊断患有晚期胰腺癌,12月下旬确诊,次年1月就去世了。这让他感到震惊,“因为她几乎没有任何征兆或症状,为什么她的癌症没有更早被发现,为什么治疗方法无效?”这些问题驱使着Poore不断地去寻找答案。
直到2017年,Poore看到《Science》杂志上的一项研究——微生物入侵大多数胰腺癌并能够分解这些患者使用的主要化疗药物。这引起了他浓厚的兴趣和猜想,细菌和病毒可能在癌症中发挥更大的作用。
念念不忘,必有回响。如今,在美国加州大学圣地亚哥医学院攻读博士的Poore师从微生物组创新中心Rob Knight教授。他们联合一个跨学科的合作小组共同开发了一种癌症诊断新方法,通过机器学习方法识别血液中存在的微生物DNA(细菌和病毒)特征,就可以诊断是否患癌以及患有哪种癌症。
这项研究结果以“Microbiome analyses of blood and tissuessuggest cancer diagnostic approach”为题于当地时间3月11日发表在《Nature》杂志上,或许会推动癌症生物学领域的重要转变。
具体来说,研究人员使用癌症基因组图谱数据库分析了18,116个肿瘤样本。这些样本来自10,481名患者,涵盖了33种不同的癌症类型,包括原发性和复发性肿瘤以及转移扩散的肿瘤。除了已知的微生物与癌症之间的联系,如人乳头瘤病毒(HPV)与宫颈癌、头颈癌之间的关系,以及梭杆菌种类与胃肠道癌症之间的关系外,研究人员还发现“新大陆”,如费氏杆菌物种与结肠癌之间的联系等。
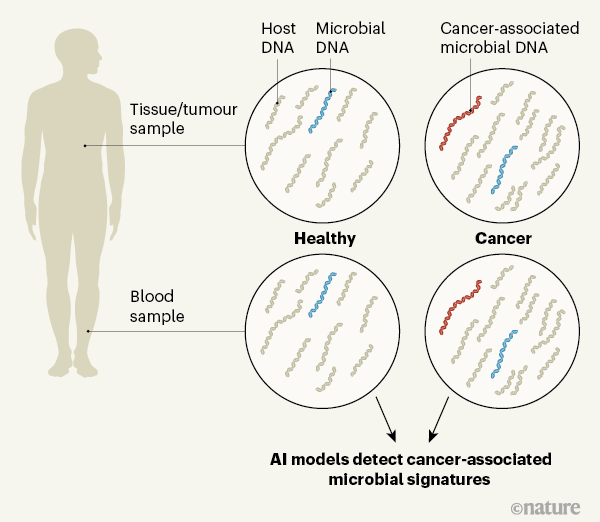
癌症的微生物特征(图片来源:Nature)
在收集了癌症样本的微生物特征之后,研究小组训练了数百种机器学习算法,以将某些微生物序列特征与特定癌症相匹配。机器学习模型能够仅使用患者血液中的微生物数据来识别患者的癌症类型。
随后,研究人员使用从59例前列腺癌、25例肺癌和16例黑色素瘤患者的血液样本中收集的数据对算法进行验证,并与69名健康个体的血液进行比较。结果显示,这些模型能以敏感性为86%识别出肺癌患者,对于无肺部疾病的个体没有出现假阳性报告;以81%的敏感性区分前列腺癌患者和肺癌患者。
目前,大多数癌症的诊断需要进行手术活检,但这种方法耗时且昂贵,而且是侵入性的。而当前的液体活检尚未能够可靠地将正常的遗传变异与真正的早期癌症区分开,罕见基因突变也很难检测。
研究人员表示,与人体循环肿瘤DNA相比,基于微生物DNA的癌症检测的一个优势是其在不同身体部位的多样性。相比之下,人类的DNA在整个身体中是基本相同的。由于不依赖于罕见的人类DNA变化,这项研究表明,与目前的液体活检相比,基于血液中微生物DNA信息或许能够在癌症早期就准确地检测出癌症的存在和类型,以及缺乏可检测基因突变的癌症。
当然,研究人员也指出,这些初步观察结果距离FDA批准其为癌症诊断检测方法,还有很长的路要走。现在需要更大、更多样化的患者群体来验证和完善机器学习算法。同时,为了加快这一商业化进程和临床应用,Knight和Poore已经申请了专利,并与合著者Sandrine Miller-Montgomery共同创立了一家名为Micronoma的公司。
原始出处:
Gregory D. Poore, et al. Microbiome analyses of blood and tissues suggest cancer diagnostic approach. Nature. Published: 11 March 2020
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言










#早诊断#
46
#Nat#
38
#微生物#
39