mBio:清华大学揭示新冠病毒奥密克戎变异株在免疫逃逸以及宿主嗜性改变的生物学机制
2023-05-09 清华大学医学院 清华大学医学院 发表于上海
该研究发现在新型冠状病毒(SARS-CoV-2)Omicron(B.1.1.529)变异株刺突蛋白中出现的大量突变增强了与不同物种ACE2受体的相互作用,并且这些变异株具有逃逸免疫反应的能力。
近日,清华大学医学院丁强课题组与中科院武汉病毒所单超课题组在《mBio》杂志发表题为《Evolution of Immune Evasion and Host Range Expansion by the SARS-CoV-2 B.1.1.529 (Omicron) Variant》的研究论文。该研究发现在新型冠状病毒(SARS-CoV-2)Omicron(B.1.1.529)变异株刺突蛋白中出现的大量突变增强了与不同物种ACE2受体的相互作用,并且这些变异株具有逃逸免疫反应的能力。该研究为深入了解新冠变异株的生物学特性提供了重要信息。
当前世界各地新冠肺炎(COVID-19)的大流行主要是由更具传染性的奥密克戎(Omicron)变异株引起的。自2021年11月24日WHO报告了首例Omicron 变异株感染病例以来1,该变异株迅速传播至全球多个国家和地区。最早出现的奥密克戎变异株为B.1.1.529,来自于B.1.1谱系,和Alpha(B.1.1.7)属于同一谱系。该变异株的刺突蛋白(Spike, S)上同时具有前4个VOC变异株Alpha(阿尔法)、Beta(贝塔)、Gamma(伽玛)和Delta(德尔塔)重要氨基酸突变位点,多达30个(图1)。不仅如此,在不断传播的过程中,奥密克戎株内部也已经进化出诸多差异颇大的支系,对疫苗研发和疫情控制带来了严峻的挑战。病毒S蛋白和细胞受体ACE2结合,从而介导病毒进入细胞,该步骤是决定物种易感性的关键步骤,也是治疗性中和抗体以及预防性疫苗发挥作用的重要靶点2,3。在新冠疫情爆发之初,丁强课题组的研究方向聚焦于探究新冠病毒的潜在宿主范围4,发现多种哺乳动物的ACE2均可作为新冠病毒的功能性受体,但是小鼠、考拉和新世界猴的ACE2不能与野生型病毒S蛋白结合支持病毒进入细胞。针对后续出现的突变株,例如 ”Cluster 5”, B.1.617.2 (Delta) 等,课题组也迅速研究了这些变异株传播能力增强的生物学机制,这些突变是否改变了S蛋白对不同物种ACE2的结合能力,以及是否会对疫苗的保护效果等重要问题5,6。在奥密克戎突变株出现之际,课题组也针对上述问题进行了探究。

图1. 新冠变异株B.1.1.529在刺突蛋白区域的突变示意图
研究者首先利用假病毒系统评估B.1.1.529对三针国产疫苗免疫者血清中和活性的敏感性,发现接种三针疫苗人群的血清对突变株的中和能力相比原始毒株有大幅下降,提示变异病毒具备免疫逃逸能力。然而,变异株病毒仍然对重组可溶性人ACE2蛋白的抗病毒活性保持敏感,这与变异株病毒刺突蛋白结合人ACE2受体能力增强的现象吻合,该结果提示重组可溶性ACE2具有广谱的抗病毒活性。
接下来,研究人员还测试了B.1.1.529刺突蛋白中的突变是否能增强对不同物种ACE2的结合能力。研究人员选取了不能和野生型病毒RBD结合的三个物种(小鼠、考拉和新世界猴)的ACE2进行研究,利用野生型以及B.1.1529的RBD蛋白,分别测定其和人、小鼠、考拉和新世界猴ACE2蛋白的结合能力,结果表明变异株RBD不仅增强了与人ACE2的结合,还获得了和小鼠ACE2结合的能力,并利用假病毒系统进一步证实了以上结果。但含有突变的RBD蛋白对考拉和新世界猴的ACE2的结合并没有表现出增强现象。另外,更加细致的功能分析表明,考拉和新世界猴ACE2的第30,31位点在不能高效介导突变S蛋白进入细胞的过程中具有重要的作用(图2)。
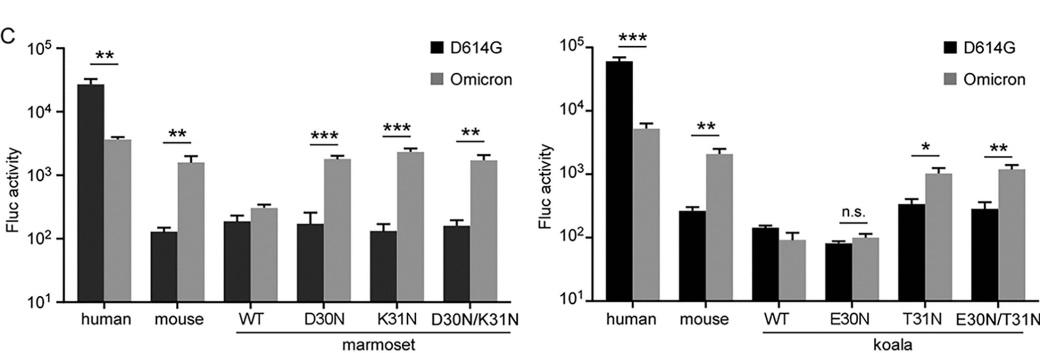
图2. 变异株假病毒进入表达不同物种ACE2细胞的能力
基于上述结果,研究人员给野生型小鼠接种了野生型和B.1.1.529突变毒株。结果表明相较与野生型,虽然感染突变株的小鼠也未导致体重下降,但是突变株在小鼠肺部的病毒RNA量和病毒载量均明显高于野生型。这说明Omicron突变株可能具备在野生型小鼠传播的能力(图3)。总之,该研究认为,Omicron突变株(B.1.1.529)与人ACE2受体结合能力增强,可以更加高效地进入细胞;此外,变异株也获得使用小鼠等其他物种ACE2的能力,具有跨物种传播的潜在风险;另外,变异株病毒也可以不同程度地逃逸人群中已经建立的免疫力,以上三种原因是导致其传播能力增强的重要因素。
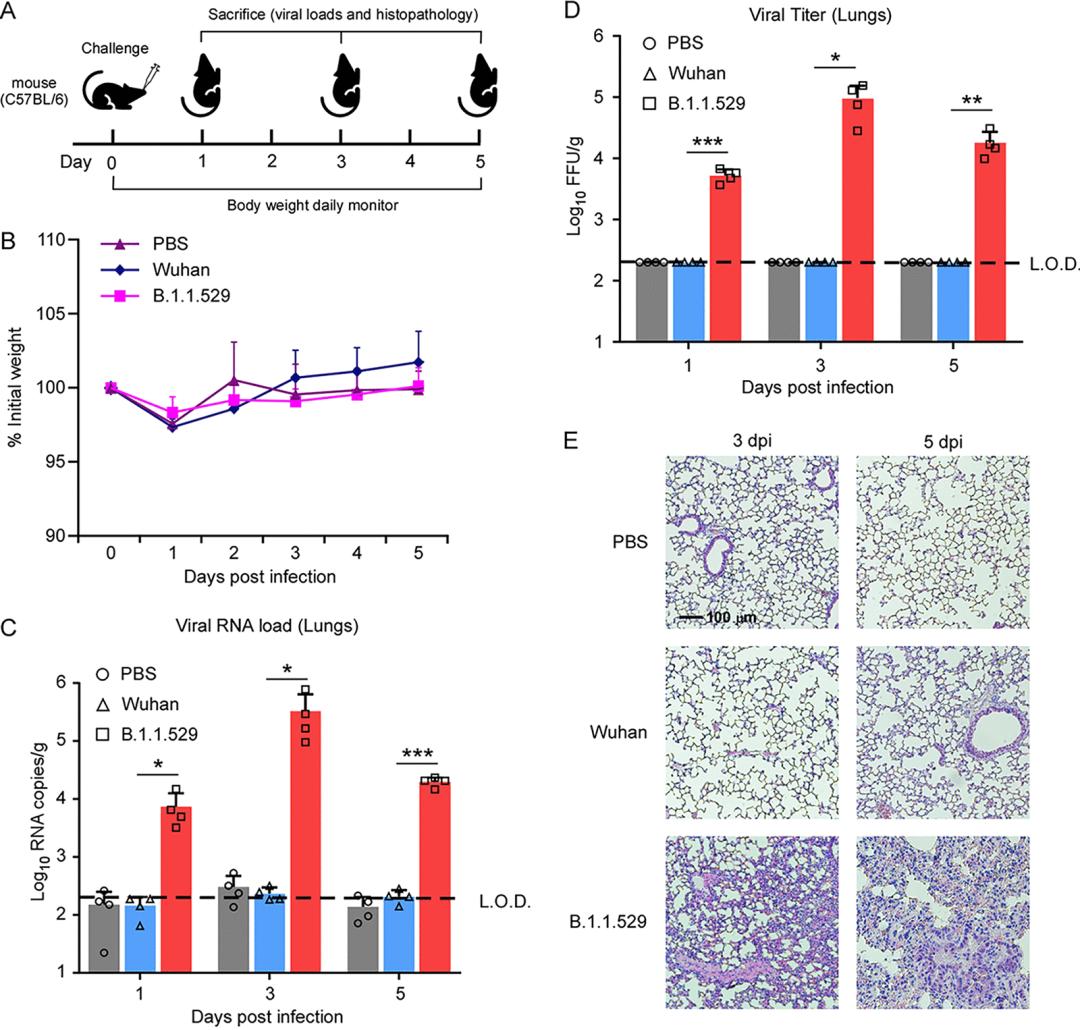
图3. Omicron突变株B.1.1.529可以感染野生型C57BL/6小鼠
清华大学医学院2018级博士生任文琳、2020级博士生张羽和武汉病毒所博士生饶具红为并列第一作者。清华大学医学院丁强研究员和武汉病毒所单超研究员为本文通讯作者。复旦大学张荣研究员、清华生命学院王新泉教授以及深圳湾实验室钟国才研究员等也对该研究提供了指导和帮助。该研究在国家自然科学基金(82272302, 82241077和32070153)的资助下完成。
参考文献:
1. Classification of Omicron (B.1.1.529): SARS-CoV-2 Variant of Concern. https://www.who.int/news/item/26-11-2021-classification-of-omicron-(b.1.1.529)-sars-cov-2-variant-of-concern
2. Lan J, Ge J, Yu J, et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor[J]. Nature, 2020, 581(7807): 215-220.
3. Wang P, Nair M S, Liu L, et al. Antibody resistance of SARS-CoV-2 variants B. 1.351 and B. 1.1. 7[J]. Nature, 2021, 593(7857): 130-135.
4. Liu Y, Hu G, Wang Y, et al. Functional and genetic analysis of viral receptor ACE2 orthologs reveals a broad potential host range of SARS-CoV-2[J]. Proceedings of the National Academy of Sciences, 2021, 118(12).
5. Ren W, Lan J, Ju X, et al. Mutation Y453F in the spike protein of SARS-CoV-2 enhances interaction with the mink ACE2 receptor for host adaption[J]. PLoS Pathogens, 2021, 17(11): e1010053.
6. Ren W, Ju X, Gong M, et al. Characterization of SARS-CoV-2 variants B. 1.617. 1 (Kappa), B. 1.617. 2 (Delta), and B. 1.618 by cell entry and immune evasion[J]. mBio, 2022, 13(2): e00099-22.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言