Nature Medicine:揭开人类大脑之谜:全新细胞图谱揭示脑细胞异质性
2024-08-06 生物探索 生物探索 发表于上海
研究人员展示了PCDH9high微胶质细胞在海马和前额叶皮质之间的基因调控差异,并阐明了细胞间的通信网络。脑细胞图谱为比较不同环境和条件下的大脑细胞提供了一个整合资源。
引言
人脑是极其复杂的器官,包含数千亿个细胞,其功能和结构多样性至今仍未完全理解。近年来,单细胞技术的快速发展极大地推动了我们对大脑细胞类型和功能的认识。然而,要深入理解大脑细胞的异质性,需要包括大量供体和多个大脑区域的研究。通过整合不同来源的单细胞数据,可以揭示稀有细胞类型和跨区域的细胞异质性。
8月2日Nature Medicine的研究报道“A brain cell atlas integrating single-cell transcriptomes across human brain regions”,介绍了一个全面的大脑细胞图谱——脑细胞图谱(Brain Cell Atlas),它结合了70个人类和103个小鼠研究中的单细胞数据,覆盖了主要发育阶段和大脑区域,总共涉及超过2630万个细胞或细胞核。通过机器学习算法,该图谱提供了一致的细胞类型注释,并展示了在人类大脑中推测的神经祖细胞(neural progenitor cells, NPCs)和高表达PCDH9的微胶质细胞(PCDH9high microglia)的识别。研究人员展示了PCDH9high微胶质细胞在海马和前额叶皮质之间的基因调控差异,并阐明了细胞间的通信网络。脑细胞图谱为比较不同环境和条件下的大脑细胞提供了一个整合资源。
自从170多年前,美国铁路工人菲尼亚斯·盖奇(Phineas Gage)的事故首次揭示了特定大脑区域对某些功能的重要性以来,研究人员通过各种研究努力理解大脑区域、细胞及其功能。尤其是在过去十年中,单细胞技术的迅猛进展加速了神经元细胞类型的发现,为区域微环境和谱系特化提供了新的见解。像人类细胞图谱(Human Cell Atlas)、人类生物分子图谱计划(HuBMAP)、BICCN和艾伦脑图谱(Allen Brain Atlas)这样的联盟积累了大量数据,提供了大规模的人类大脑细胞参考图谱。
尽管有一些例外,目前大多数研究都局限于单一区域、小部分细胞或特定疾病,并通过分离的数据集进行存档,这导致了一些与疾病和发育过程相关的细胞类型或细胞状态尚未被探索。整合来自各种数据集的数据可能丰富这些细胞类型的信息,从而带来重要发现,包括不同年龄段的神经发生(neurogenesis)、稀有细胞的发现、区域异质性及细胞类型对神经退行性疾病的贡献。
脑细胞图谱整合了70项研究中的1130万个人类细胞和103项研究中的1500万个小鼠细胞,覆盖了几乎所有主要的大脑区域,包括健康和疾病状态。该研究展示了脑细胞图谱在发现成人中的推测神经祖细胞和理解微环境驱动的微胶质细胞差异中的应用。这个图谱将作为研究大脑细胞和功能的重要资源,提升我们对神经元过程和神经退行性疾病的理解。

人类大脑是由无数种细胞组成的复杂器官,其功能和结构的多样性至今仍未完全了解。单细胞RNA测序(single-cell RNA sequencing, scRNA-seq)技术的快速发展,为研究大脑细胞的异质性和特定区域的功能提供了前所未有的机会。然而,由于个体差异、不同研究的技术方法和数据质量的差异,整合多个数据集以获得一致的细胞类型注释仍然面临巨大挑战。该研究通过整合70项人类和103项小鼠大脑研究中的单细胞数据,创建了一个全面的大脑细胞图谱,为研究大脑的复杂性和多样性提供了宝贵资源。
研究人员收集了70个人类大脑研究和103个小鼠大脑研究的单细胞转录组数据。数据来自GEO、CELLxGENE、UCSC基因浏览器、ArrayExpress等数据库 。对原始数据进行质量控制,包括去除低质量细胞、双重细胞(doublets)和线粒体含量过高的细胞 。
使用七种监督机器学习方法(ACTINN, scArches, CHETAH, scmap, SingleCellNet, SingleR, scPred)以及自建工具scAnnot对细胞进行重新注释 。通过scANVI模型进行多层次细胞类型注释,从而在不同分辨率下训练和应用机器学习模型,识别主要细胞类型和特定细胞类型 。
为了解决批次效应和技术噪声问题,研究人员采用了整合后的数据进行差异表达分析 。使用edgeR程序对汇总计数数据进行分析,模型中包括了生物因素和协变量以减少批次效应的影响 。
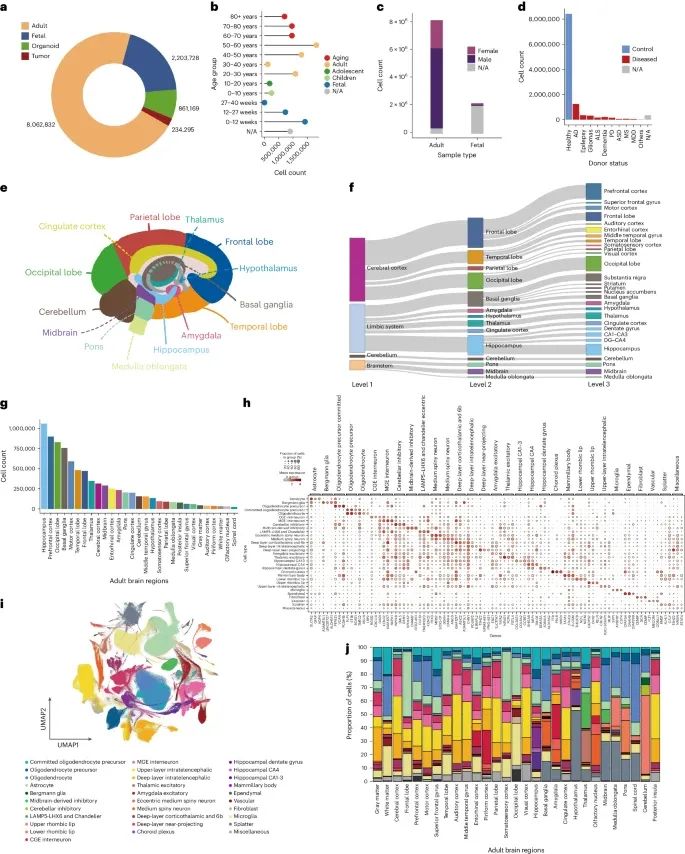
脑细胞图谱中样本的多样性和广泛覆盖(Credit: Nature Medicine)
样本类型的比例:脑细胞图谱中的样本类型按比例分布,成人样本占80.6%,胎儿样本占22.0%,类器官样本占8.6%,肿瘤样本占2.3%。
细胞数量与年龄分布:不同年龄段的细胞数量分布,从胎儿期的6周到成人80岁以上,详细展示了不同发育阶段和年龄组的细胞数量。
供体性别比例:成人样本中,女性供体占24.6%,男性供体占70.7%,性别信息不明的占少数。胎儿样本中,女性供体略多于男性供体,性别不明的占多数。
健康与疾病状态的细胞数量:大多数样本来自健康供体,少数样本来自各种疾病状态,包括阿尔茨海默病、癫痫、胶质瘤、肌萎缩侧索硬化症、重度抑郁症、自闭症、帕金森病和多发性硬化症。
主要大脑区域的细胞数量分布:样本覆盖了主要的大脑区域,包括额叶、顶叶、枕叶、颞叶、小脑、脑干和边缘系统等。海马区样本数量最多,其次是前额叶皮质、枕叶和基底神经节。
大脑细胞图谱概览
该资源包括来自14个主要大脑区域和30个亚区域的1130万个成人细胞和来自103个小鼠研究的1500万个细胞 。
人类大脑数据按样本来源分类为成人(8062832个细胞)、胎儿(2203728个细胞)、类器官(861169个细胞)和脑肿瘤(234295个细胞) 。
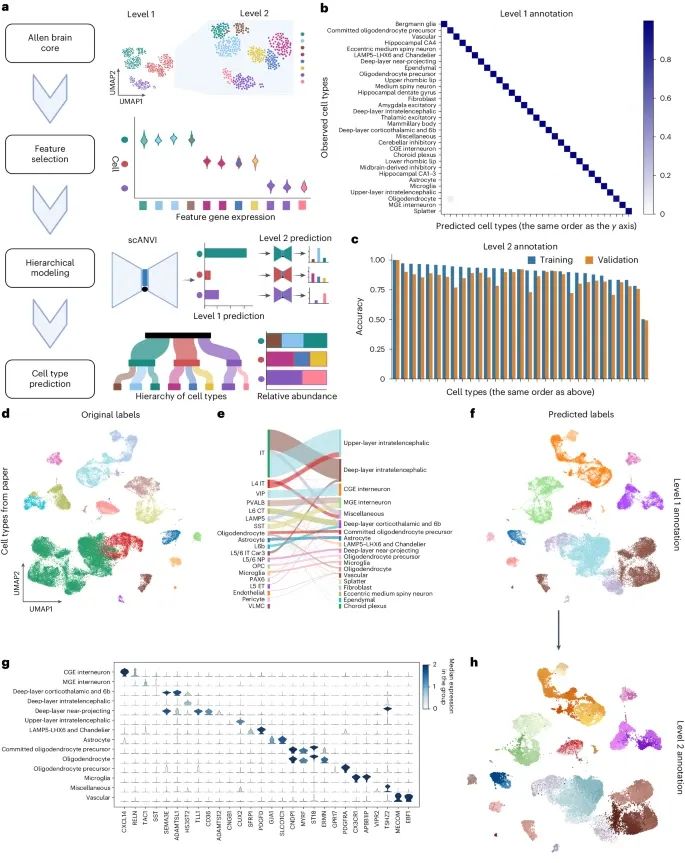
scAnnot工具在细胞类型注释中的高准确性和多层次分类能力(Credit: Nature Medicine)
scAnnot工具的层次细胞注释模型:介绍了scAnnot工具的层次分类模型。该模型基于scANVI算法,将细胞按层次进行分类。第一层分类将细胞分为广义细胞类型,第二层则进一步细化分类。
第一层细胞类型预测的准确性:展示了第一层细胞类型预测的热图,行表示已报道的细胞类型,列表示scAnnot预测的细胞类型。颜色越深表示预测准确性越高。
第二层细胞类型的训练和验证准确性:用条形图显示第二层细胞类型的训练和验证准确性。X轴表示广义细胞类型,Y轴表示预测准确性,蓝色和橙色条分别代表训练和验证的准确性。
已报道和预测的第一层细胞类型的UMAP可视化:使用UMAP可视化已报道和scAnnot预测的第一层细胞类型。颜色表示不同的细胞类型。
特征基因的表达水平:展示了不同细胞类型的特征基因表达水平的叠加小提琴图。Y轴表示基因表达水平,X轴表示基因名称,不同行表示不同细胞类型。
第二层scAnnot预测的细胞类型UMAP可视化:使用UMAP可视化第二层scAnnot预测的细胞类型,展示了更细化的细胞类型分类。
成人海马区的推测神经祖细胞(NPCs)
通过标记基因识别、免疫染色、基因模块评分、轨迹推断和跨物种比较等方法,研究人员推测在成人海马区存在神经祖细胞 。
推测的NPCs表达增殖神经祖细胞的分子标志物,例如SOX2、NES、ASCL1等 。
高表达PCDH9的微胶质细胞(PCDH9high Microglia)
在整合的数据中识别出在前额叶皮质和海马区高表达PCDH9的微胶质细胞,表现出促炎症表型和吞噬髓鞘碎片的潜力 。
这些PCDH9high微胶质细胞在基因表达和功能上与其他微胶质细胞存在显著差异 。
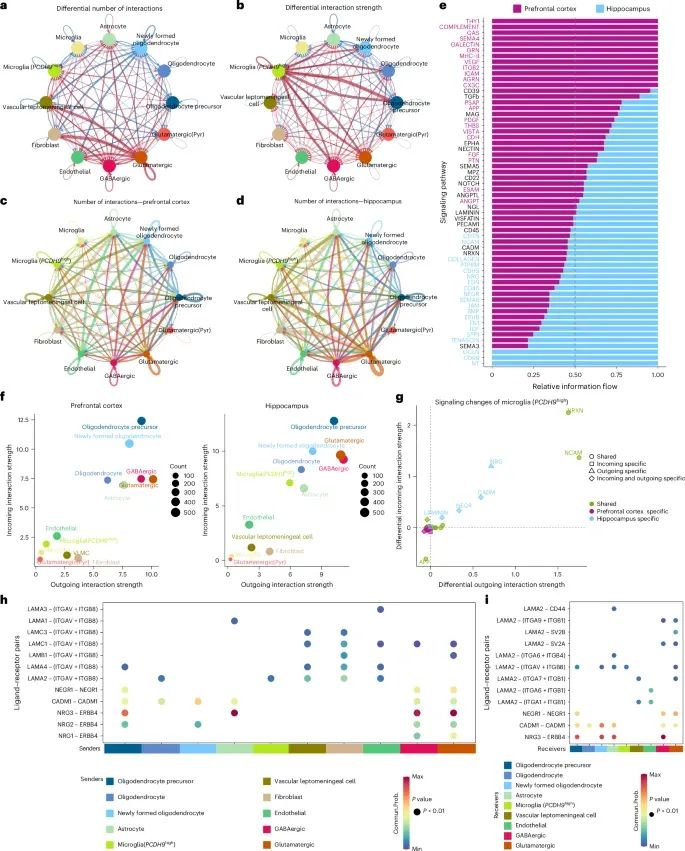
前额叶皮质和海马区的细胞间通信模式存在显著差异,这些差异可能与各自的功能和微环境特性相关(Credit: Nature Medicine)
细胞间通信网络示意图:该图展示了前额叶皮质和海马区内神经发生微环境中细胞间通信网络的概览。通过CellChat程序分析了细胞间可能的配体-受体相互作用。
通信通路的数量和分布:展示了60条通信通路(980个基因)参与了神经细胞环境的细胞-细胞通信网络构建。其中包括45条保守的通信通路,12条前额叶皮质特异的通路和3条海马特异的通路。
前额叶皮质和海马区的细胞间通信强度:使用二维空间图展示了前额叶皮质和海马区内细胞间通信的发送和接收强度的变化。显示了在两个脑区内,不同细胞类型之间通信强度的差异。
前额叶皮质和海马区特异的信号通路:分析表明,海马区的微胶质细胞(PCDH9high)显示了特异性的信号变化,主要体现在NRG、CADM、NEGR和Laminin通路的特异变化中。
配体-受体对的具体细胞发送和接收情况:详细列出了在海马区,十个发送细胞通过NRG、CADM、NEGR和Laminin通路与微胶质细胞(PCDH9high)进行通信。这些发送细胞包括少突胶质细胞前体细胞、成纤维细胞、内皮细胞、GABA能神经元和谷氨酸能神经元等。
前额叶皮质和海马区的差异信号传递:展示了两脑区在细胞-细胞通信网络中差异信号传递的具体变化。前额叶皮质中的微胶质细胞(PCDH9high)表现出高度的促炎性和吞噬状态,而海马区的这些细胞则更多地参与调节突触可塑性
该研究展示了一个覆盖多个大脑区域和发育阶段的大脑细胞图谱,提供了一致的细胞类型注释。这一图谱揭示了成人海马区推测神经祖细胞和区域特异性微胶质细胞的异质性,为未来研究大脑功能和疾病机制提供了重要资源。
整合大规模数据集的过程中,处理批次效应和减少技术噪声仍然是主要挑战。然而,通过建立一个统一的大脑细胞参考图谱,我们能够更好地理解大脑细胞的多样性和区域特异性,为探索未解之谜和开发新疗法提供了基础。
参考文献
Chen X, Huang Y, Huang L, Huang Z, Hao ZZ, Xu L, Xu N, Li Z, Mou Y, Ye M, You R, Zhang X, Liu S, Miao Z. A brain cell atlas integrating single-cell transcriptomes across human brain regions. Nat Med. 2024 Aug 2. doi: 10.1038/s41591-024-03150-z. Epub ahead of print. PMID: 39095595.
https://www.nature.com/articles/s41591-024-03150-z
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#脑细胞# #细胞图谱#
54