PNAS:利用噬菌体对微生物群落中特定细菌进行碱基编辑
2022-11-11 王聪 “生物探索”公众号
两种工程噬菌体——T7噬菌体和λ噬菌体,都能够向大肠杆菌宿主递送有效载荷,这些有效载荷表达细菌荧光基因,并操纵细菌对抗生素的耐药性。
近日,北卡罗莱纳州立大学的研究人员在《美国国家科学院院刊》(PNAS)上发表了题为:CRISPR-based engineering of phages for in situ bacterial base editing 的研究论文。

论文通讯作者、北卡罗莱纳州立的 Rodolphe Barrangou 教授表示,这项研究表明,可以对微生物群落系统中的特定细菌进行基因编辑,而不影响其他微生物,这项研究可以进一步开发并应用于任何复杂的微生物群落,有望转化为更好的植物健康和更好的胃肠道健康环境。
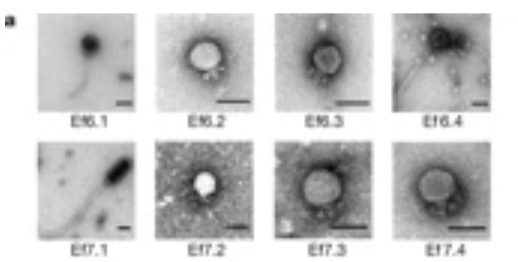
首先,研究团队研究了T7噬菌体和λ噬菌体是否可以通过工程设计将CRISPR系统递送给大肠杆菌。
像λ噬菌体这样的噬菌体可以将它们的遗传物质整合到宿主细菌的遗传物质中,并与宿主细菌进行协同复制,这种不会因为噬菌体感染而导致细菌裂解的现象被称为溶原现象(lysogeny)。
这项研究显示,两种工程噬菌体——T7噬菌体和λ噬菌体,都能够向大肠杆菌宿主递送有效载荷,这些有效载荷表达细菌荧光基因,并操纵细菌对抗生素的耐药性。
研究团队表示,病毒非常擅长递送基因,而这项研究使用噬菌体,将CRISPR系统递送给细菌,这颇具讽刺意味,因为CRISPR本是细菌用来对抗噬菌体的防御系统。而这项研究反过来用噬菌体递送CRISPR来编辑细菌,病毒在此时发挥了注射器一样的作用。
研究团队使用λ噬菌体将胞嘧啶碱基编辑器(CBE)递松到大肠杆菌中,以高灵敏度和精确度改变大肠杆菌DNA中的单个碱基。
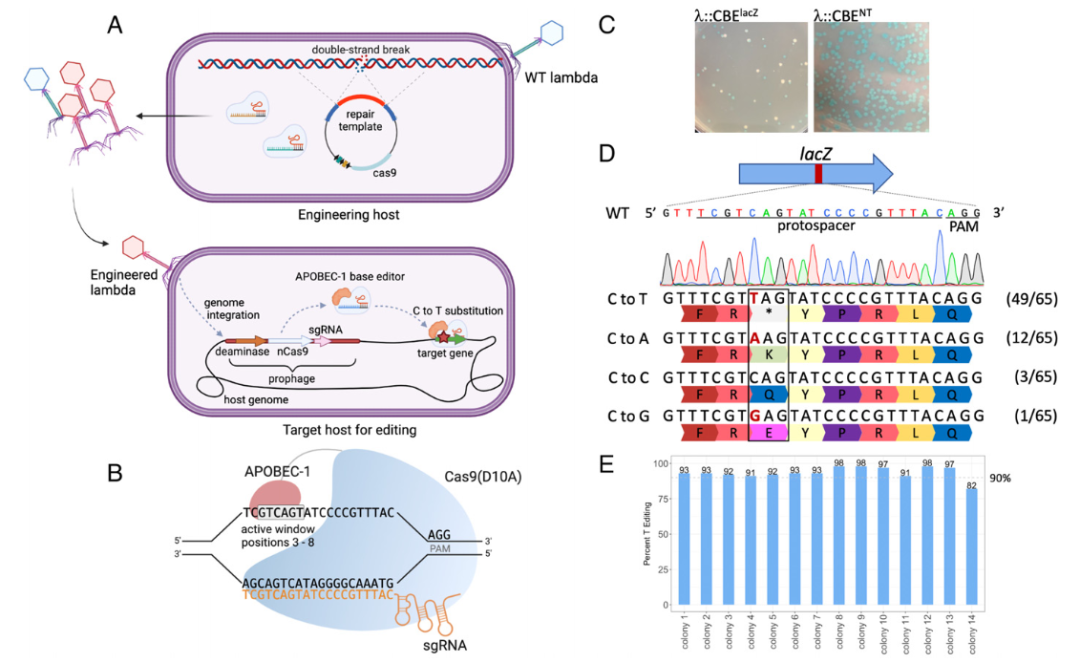
论文的第一作者 Matthew Nethery 博士表示,这项研究使用碱基编辑器(Base Editor)作为大肠杆菌基因的一种可编程开关。使用这样的系统,可以对基因组进行高度精确的单碱基编辑,而不需要CRISPR-Cas系统那样导致双链DNA断裂,从而导致潜在风险。
在EcoFab的合成生态系统中,研究团队加入了三种不同类型的细菌,以研究这些工程噬菌体是否可以在生态系统中特异性定位大肠杆菌。实验结果显示,工程λ噬菌体可以有效地附着在大肠杆菌上并编辑其基因组。
研究团队使用工程噬菌体在试管和目标细菌存在的微生物群落中进行原位递送的CRISPR-Cas基因编辑。使用合成土壤构建了包括一系列细菌的微生物群落模型。了解土壤根际相互作用对确保粮食资源的可持续性非常重要。这项研究为利用土壤微生物群落控制植物伴生菌的组成和功能奠定了基础。这些发现可以帮助设计生态系统,以促进植物的生长和健康,这对可持续农业至关重要。
研究团队表示,未来的实验中,将使用其他与土壤相关的细菌来测试这种噬菌体CRISPR技术。
原始出处:
CRISPR-based engineering of phages for in situ bacterial base editing. PNAS, 2022.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言