Nature Communications:选择性SMARCA2-PROTAC
2022-12-25 精准药物 精准药物 发表于安徽省
作者发现了一种全新的选择性SMARCA2-PROTAC药物分子,在SMARCA4mut模型中显示出强效的体外生长抑制和体内疗效
2022年11月10日,美国Genentech公司的Robert L. Yauch团队在Nature Communications上发表了题为《Selective PROTAC-mediated degradation of SMARCA2 is efficacious in SMARCA4 mutant cancers》的文章。作者在文章中报道了一种强效、选择性的SMARCA2-PROTAC, A947。它在缺乏选择性SMARCA2/4结合的情况下,实现了选择性SMARCA2降解,并且在SMARCA4mut模型中,与野生型相比具有强效的体外生长抑制和体内疗效。全局泛素化图谱和蛋白质组学分析显示用A947处理未出现相关的脱靶效应,这为含有SMARCA4mut的肿瘤患者提供了新的潜在治疗机会。
SWI/SNF是一种多亚基染色质重塑复合体,能够促进染色质重构,以调节关键的细胞过程,包括转录调节和DNA修复。其催化功能由两种相互排斥的ATP解旋酶SMARCA2/4赋予。SMARCA2/4具有很强同源性,除了高度保守的ATPase结构域(93%的同源性)外,这两种蛋白质都含有一个保守的溴主结构域(96%的同源性)(BD),可以与乙酰化染色质相互作用。SMARCA4功能缺失突变在多种恶性肿瘤亚群中富集,纯合突变主要发生在非小细胞肺癌(NSCLC) 中。SMARCA2蛋白对于SMARCA4功能缺失的细胞具有一定的功能补偿性,在成年小鼠中,同时敲除SMARCA2和SMARCA4基因将导致小鼠死亡。由于SMARCA2/4具有很强同源性,因此抑制剂往往选择性不佳,如何实现SMARCA2的选择性抑制成了科学家们研究的热点。蛋白水解靶向嵌合体(Proteolysis targeting chimeras, PROTACs)是一种新兴的治疗方式,它依赖形成三元复合物发挥蛋白质的降解作用,与传统的小分子抑制剂相比,更具有选择性,并且其通过非选择性的配体实现选择性的蛋白降解已经被证实。
作者将一个能够结合SMARCA2/4和PBRM1的溴域的小分子配体(5th BD)与VHL配体连接起来合成了PROTAC分子 A947 (图1a)。A947与SMARCA2和SMARCA4的溴域结合亲和力没有差异(图1b)。(SMARCA2 Kd =93nM, SMARCA4 Kd = 65nM) 在SW1573细胞中,A947潜在地降解SMARCA2,DC50为39 pM, 在10nm时达到96%的最大降解(图1c,d)。相比之下,需要28倍浓度A947才能在SMARCA4上实现DC50(1.1nM),在接近100nM时实现92%的最大降解。这种降解选择性的程度与所评估的SMARCA2/4的特定亚型无关(图1e)。此外,A947对降解PBRM1表现出类似的选择性(图1e,1 d)。通过竞争性rescue实验,作者发现A947对SMARCA2的降解依赖于泛素蛋白酶体途径(图1f)。全局泛素图谱和蛋白质组学分析进一步验证了A947在高浓度下降解这些靶蛋白具有高度特异性(图2a、b)。
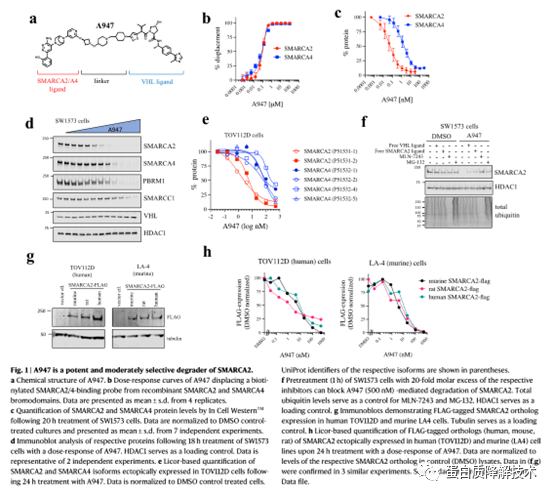
图 1

图 2
接下来作者评估了A947对细胞增殖的影响。在SMARCA4 mut NCI-H1944细胞中,A947能够剂量依赖性地抑制其生长 (图3a)。在多种SMARCA4 mut肺癌模型(图3b, c)以及两种缺乏SMARCA2/4表达的细胞系中,SMARCA4 mut肺癌细胞系对A947治疗最敏感,整个细胞系的中位IC 50为7nM,SMARCA4 WT细胞对A947的敏感性显著降低,中位IC 50为86nM。对缺乏SMARCA2/4表达的细胞生长无影响。a947介导的降解主要导致SMARCA4 mut模型的G1阻滞,而在对照细胞系中没有观察到(图3d)。在转录水平上,A947介导的SMARCA2降解主要导致SMARCA4 mut细胞的转录抑制,这与SMARCA2作为染色质调节器的作用一致(图3e)。
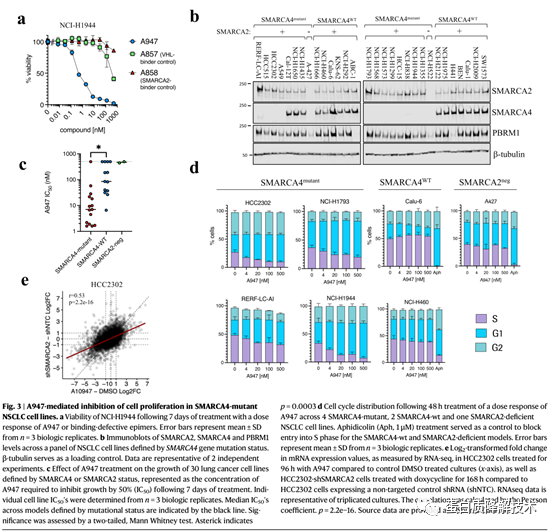
图 3
接下来,作者初步评估了在SMARCA4 mutHCC515异种移植体内单次静脉注射40mg / kg A947后的药效学(PD)效应(图4a),静脉注射A947可使肿瘤SMARCA2蛋白水平迅速降低(4h降低96%),并在24h时达到最大降低。随后,作者采用每隔一周40mg / kg静脉注射A947的给药方案,对两种不同的SMARCA4 mut肺癌异种移植模型HCC515和HCC2302进行药效学研究(图4b, c)。在两种模型中均观察到肿瘤生长的统计学显著降低,HCC515模型的生长几乎完全抑制,HCC2302模型的肿瘤生长抑制达到60%。研究结束时还测量了肿瘤药效学和生物标志物反应;给药后24小时。A947治疗导致两种模型中肿瘤SMARCA2蛋白水平下降95%以上,但在HCC515模型中观察到KRT80转录的抑制略强(图4d)。为了确定SMARCA4 mut模型中的肿瘤生长抑制是否是由于SMARCA2降解引起的肿瘤细胞自主效应,作者使用A947评估了在SMARCA4wtCalu-6异种移植物模型中的肿瘤生长抑制 (图4f)。A947在SMARCA4wt Calu-6异种移植中没有导致肿瘤生长抑制,尽管实现了超过95%的SMARCA2蛋白降解(图4g)。SMARCA4和PBRM1在用药后24小时出现中度降解,分别降低58%和57%。综上所述,这些数据支持SMARCA2降解的肿瘤细胞内在效应,并为合成的致死相互作用提供了药理学支持。
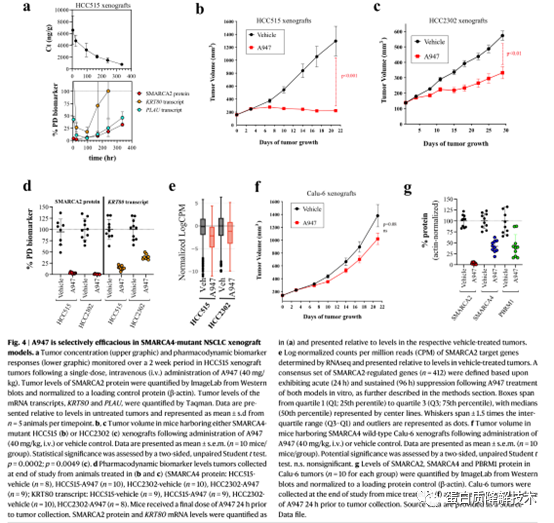
图 4
为了评估药物的联合效应,作者筛选了723种实验和临床阶段的药物与A947联合使用,在4种SMARCA4 mut肺癌细胞系中评估联合效应(图5a)。MCL1抑制是唯一一种与A947在多个SMARCA4 mut模型(3个模型)表现出强烈敏化作用的组合,在多种情况下,A947介导的SMARCA2降解表现出与MCL1抑制的协同相互作用,在SMARCA4 WT模型中未发现这一协同作用。

图 5
综上,作者发现了一种全新的选择性SMARCA2-PROTAC药物分子,在SMARCA4mut模型中显示出强效的体外生长抑制和体内疗效,并且可以与MCL1抑制剂联合使用增强抗肿瘤效果,为SMARCA4mut癌症患者提供了一种潜在的临床治疗方法。
服务:
本公众号免费接受科研团队/单位的研究进展、研发故事等非商业/非盈利目的投稿,及免费发布科研团队的招聘广告等,欢迎投稿。
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言











好文章,谢谢分享。
75