深圳市第三人民医院研究团队《自然·通讯》:系统揭示猴痘病毒的进化轨迹和特征
2024-09-03 BioMed科技 BioMed科技 发表于上海
系统揭示了猴痘病毒Clade IIb的进化轨迹和特征,首次鉴定了新的C.1.1谱系。
2024年8月28日,深圳市第三人民医院(南方科技大学第二附属医院)/深圳国家感染性疾病临床医学研究中心卢洪洲/刘映霞/杨扬教授团队在Nature Communications杂志在线发表了题为“Evolutionary trajectory and characteristics of Mpox virus in 2023 based on a large-scale genomic surveillance in Shenzhen, China”的研究论文,系统揭示了猴痘病毒Clade IIb的进化轨迹和特征,首次鉴定了新的C.1.1谱系。

猴痘病毒(Mpox virus,MPXV),是一种有包膜的双链DNA病毒,属于痘病毒科、正痘病毒属的成员,1958年首次分离自发热的猕猴体内,1970年在刚过首次报道感染人感染病例。2021年之前,猴痘病毒主要流行区域一直局限于中非和西非国家,这些地区偶发的猴痘疫情通常与感染动物的直接接触有关。2022年5月份以来,猴痘病毒波及全球的121个国家和地区流行,累计确诊病例数已超过10万例(截至2024年7月31日)。自1980年世界卫生组织(WHO)宣布天花病毒被人类彻底灭绝之后,猴痘病毒已成为全球公共卫生领域威胁最大的正痘病毒。猴痘病毒可以分为两个主要分支,Clade I 的猴痘病毒引起的病情较重,病死率可达10.6%;而Clade II 猴痘的症状较轻,全球病死率约为0.22%。近期非洲疾控中心报道了自2024年以来,非洲地区新增了14,250例猴痘患者,较去年同期激增160%。同时WHO提示Clade I分支已经演化出Clade Ib分支,并在非洲大规模传播,WHO于2024年8月14日再次宣布猴痘疫情为国际关注的突发公共卫生事件(PHEIC)。因此,阐明猴痘病毒的进化轨迹和特征,能够为我们进一步理解其传播和致病机制提供重要参考信息。
自2023年6月以来,深圳地区开始出现猴痘病毒的持续本土流行。本研究团队迅速建立了临床研究队列,纳入了2023年6月9日至10月12日期间来院就诊的的92例猴痘感染患者,通过全基因组扩增测序获得了92个高质量的MPXV全基因组序列,进一步结合系统进化分析阐明了深圳地区流行的猴痘病毒的可能来源,阐明了猴痘病毒Clade IIb的进化路径和特征。
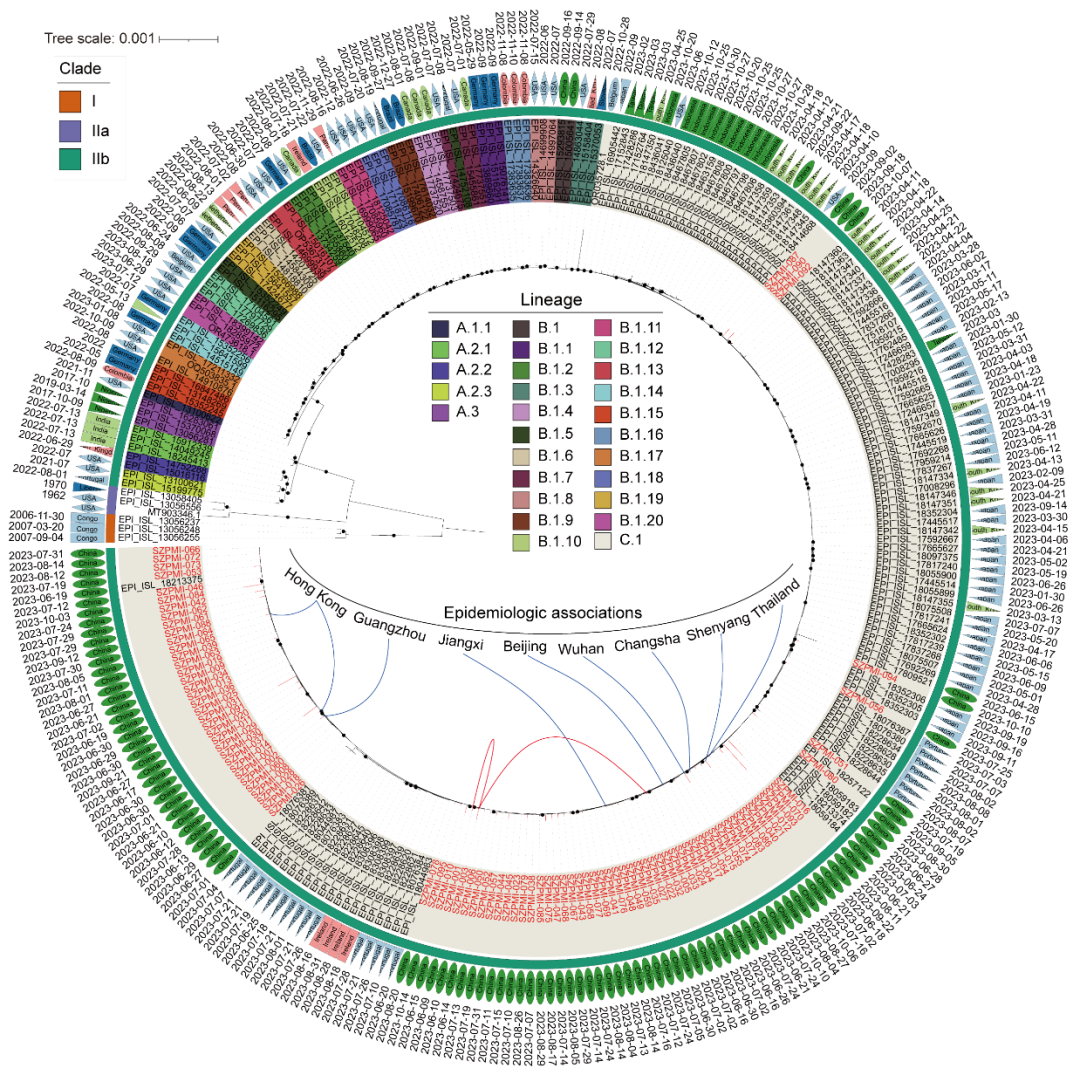
2023年深圳本土92株猴痘病毒全基因组进化分析
本次研究纳入了92例病例,分别占广东省和深圳市同期报告病例数的30%和66%。人口学特征显示,所有患者均为男性,中位年龄为30岁,56.5%的患者同时确诊了人类免疫缺陷病毒(HIV)。流行病学调查显示,其中95.7%存在男男性行为(MSM)。值得注意的是,患者全身多个部位均有皮肤病变,62.2%的患者有生殖器病变,提示猴痘病毒通过性接触传播的风险较高。
系统发育分析结果显示,92株深圳MPXV都位于谱系C.1内,除2022年重庆首次报道的毒株外,2023年中国测序的其他4株猴痘病毒也均属于C.1谱系。基因组进化关系提示深圳地区猴痘病毒可能3个不同来源地(韩国、日本和葡萄牙)。尽管仅有2对患者报告了流行病学关联,但是全基因组系统进化分析显示深圳地区猴痘病毒基因组聚集成簇,提示猴痘病毒在深圳地区MSM人群中已经建立了至少2个稳定的传播链。
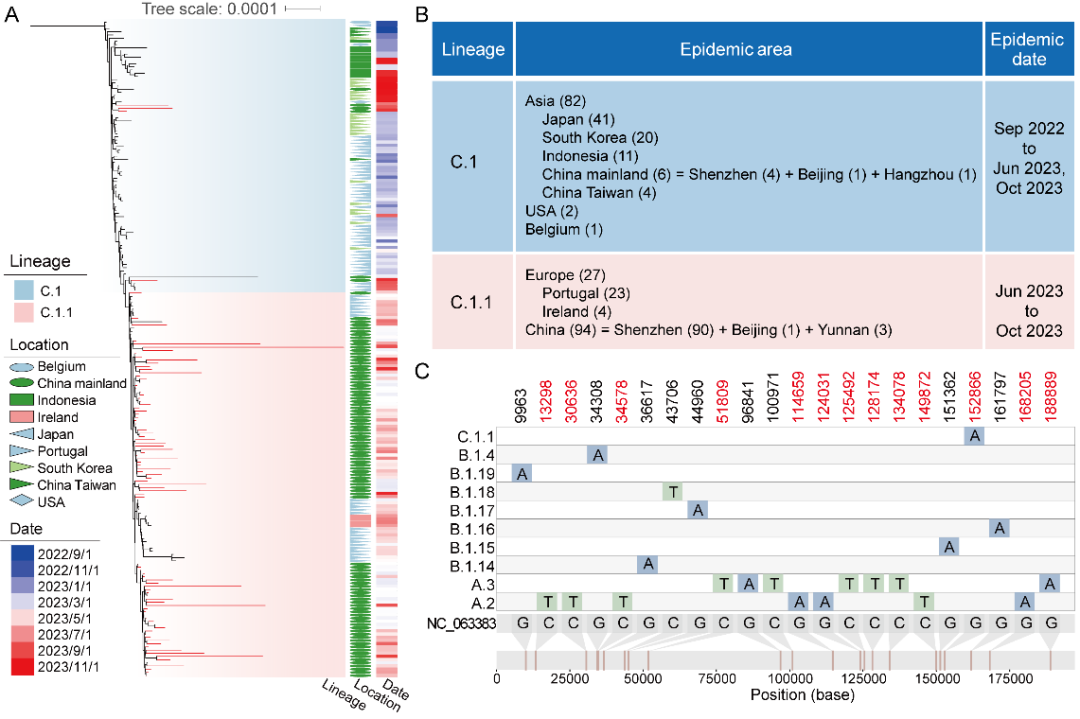
2023年猴痘病毒新谱系C.1.1的出现
我们进一步对MPXV进化树中的C.1谱系进行了深入分析,发现C.1谱系从流行地域和流行时间可以进一步分为2个不同的亚谱系:C.1和C.1.1。其中C.1谱系的流行区域主要在亚洲,包括日本、韩国、印尼以及中国。此次深圳猴痘病毒中有4例属于C.1分枝,前期文献报道的中国北京和杭州的2例猴痘病毒也属于此谱系。同时,C.1谱系的主要流行时间从2022年9月至2023年6月。而C.1.1谱系主要流行于欧洲的葡萄牙和爱尔兰,以及中国深圳、北京、云南地区,此次我们报道的深圳猴痘病毒大部分属于C.1.1谱系。从时间上看,C.1.1谱系病毒的流行时间集中在2024年的6-10月。进一步,我们绘制了Clade IIb猴痘病毒的核苷酸突变图谱,突变图谱的聚类结果与系统发育树中的谱系分类结果一致,进一步支持了系统发育关系的稳定性以及新谱系C.1.1的出现。同时,我们在C.1.1谱系中鉴定了谱系特异的突变位点G152866A,其会导致OPG180蛋白 (A50R, DNA连接酶) 的Asp59Asn突变,为新谱系C.1.1的出现提供了进一步地支持。
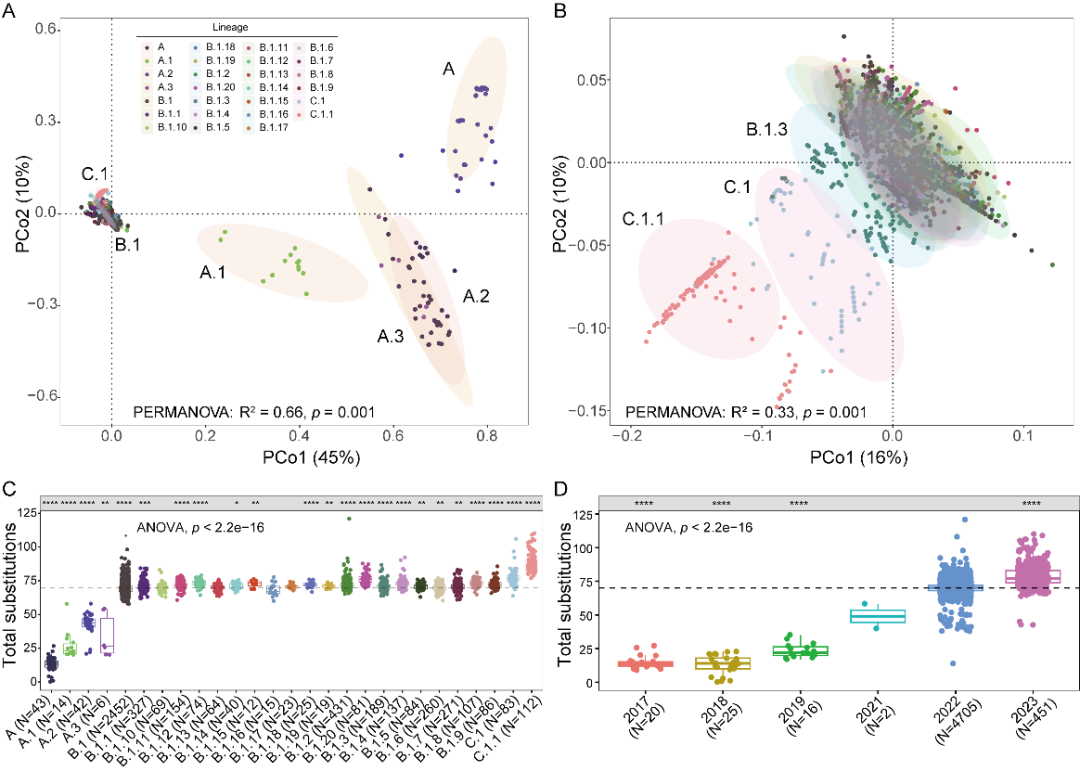
猴痘病毒Clade IIb基因组的演化历史
随后,我们解析了Clade IIb猴痘病毒在人群中持续传播的演化动态,特别是新出现的C.1.1谱系。使用PCoA对5220个Clade IIb基因组中提取的7432个核苷酸变异数据进行降维分析,结果表明不同MPXV谱系间的突变差异显著(R2 = 0.66, p= 0.001)。整个Clade IIb猴痘病毒大致可分为四个不同的进化过程,包括最早形成的A谱系、源自A谱系的A.2和A.3谱系、源自A谱系的A.1谱系、以及源自A.1谱系的B.1和C.1谱系。特别是,B.1谱系的分化显示出一个新的进化方向。随后,从B.1谱系分化出来的21个B.1亚谱系表现出明显的聚类特征。值得注意的是,C.1.1谱系与其他谱系显著分离,这表明它可能经历了另一个重要的进化事件。进一步,核苷酸突变数的全局比较也显示出与PCoA结果相对应的进化过程,也即A谱系中的突变数量显著低于B.1谱系,C.1.1谱系的突变数量显著高于Clade IIb的平均水平。此外,我们观察到,随着时间的推移,MPXV中核苷酸突变数量的仍在持续增加。
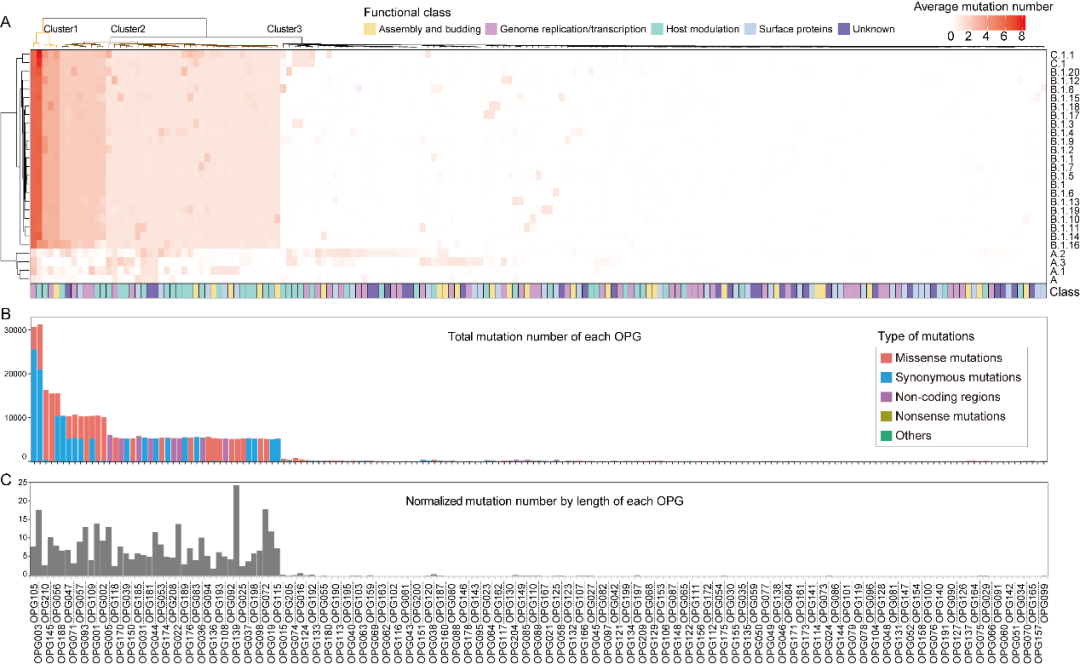
猴痘病毒Clade IIb的蛋白突变图谱
我们进一步分析了猴痘病毒的蛋白突变图谱,发现在MPXV的进化过程中,不同的蛋白质可能承受了不同的选择压力。27个谱系在蛋白质突变谱上的聚类关系与系统进化关系一致:谱系A中的蛋白突变较少,与其他谱系明显分离,谱系B和C中的蛋白突变显著地增加。随着B谱系在人群中的传播,Cluster 1和Cluster 2两个蛋白簇中的蛋白累积的蛋白突变较多,其中许多蛋白与宿主调节功能相关,这表明这些蛋白在MPXV的人类传播中起重要作用。值得注意的是,我们的研究首次揭示了在C.1.1谱系中出现的C21062T突变导致N2L基因的转录起始中断。N2L基因的突变已被证明可以降低痘苗病毒的毒力,因而其也可能影响猴痘病毒C.1.1谱系的毒力与适应性。这些结果表明,MPXV在其持续进化过程中积累了与毒力和表面抗原蛋白相关的突变,需要对其致病性和传播性的潜在变化保持警惕。进一步研究这些突变的生物学意义和潜在影响,将有助于更好地理解MPXV的进化机制,并为流行病监测和干预策略提供科学依据。
深圳市第三人民医院(南方科技大学第二附属医院)/深圳国家感染性疾病临床医学研究中心卢洪洲教授、刘映霞教授和杨扬教授为本文共同通讯作者,张圣杰博士、王福祥教授、彭运为本文的共同第一作者。
原文链接:
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言














#猴痘病毒# #Clade IIb#
65