Nat Commun:邵志敏/江一舟/杨文涛/徐军团队开发sc-MTOP方法解析乳腺癌肿瘤生态系统的多样性及临床相关生态亚型
2023-11-24 测序中国 测序中国 发表于上海
基于该数据集,研究团队全面剖析了乳腺癌生态系统的表型多样性,发现了与不同分子特征和患者预后相关的四种乳腺癌生态型,并揭示了与临床相关的生态系统特征。
肿瘤越来越被认为是一个由肿瘤细胞、免疫细胞和基质细胞以及复杂的细胞间相互作用组成的“生态系统”。肿瘤的预后和治疗反应不仅取决于肿瘤细胞的特性,还与其他细胞成分及其与肿瘤细胞的相互作用有关。解析肿瘤生态系统的组成及不同细胞类型之间的关系,对理解肿瘤生物学和提高肿瘤治疗水平具有重要意义。
目前,组织病理学是癌症诊断的“金标准”。除诊断信息外,病理切片还提供了丰富的肿瘤生态系统信息。近年来,数字病理学和计算机图像分析的出现允许科研人员从病理全载玻片图像(WSI)中高通量表征肿瘤生态系统。但迄今仍缺乏在单细胞水平上表征肿瘤生态系统中每种细胞类型表型多样性的综合图谱。
复旦大学上海肿瘤中心邵志敏、江一舟、杨文涛与南京信息工程大学徐军团队合作在Nature Communications上发表了题为“Single-cell morphological and topological atlas reveals the ecosystem diversity of human breast cancer”的文章。研究团队提出单细胞形态和拓扑分析(sc-MTOP)框架,通过提取单个细胞的细胞核形态和细胞间空间关系特征来表征肿瘤生态系统,并构建了包含来自637例乳腺癌WSI的4.1亿个细胞的单细胞图谱数据集,分别剖析了肿瘤细胞、炎症细胞和基质细胞的表型多样性。基于该数据集,研究团队全面剖析了乳腺癌生态系统的表型多样性,发现了与不同分子特征和患者预后相关的四种乳腺癌生态型,并揭示了与临床相关的生态系统特征。

文章发表在Nature Communications
sc-MTOP框架以数字病理学为基础,能够在单细胞水平上表征肿瘤生态系统(图1)。sc-MTOP框架以H&E染色的病理WSI(40×)作为输入,通过Hover-Net进行同步核分割和分类;然后对单个肿瘤细胞、炎症细胞和基质细胞进行特征分析。基于细胞核轮廓提取细胞核形态学特征和纹理特征;并设计了基于图的方法(多层成对图)对不同细胞类型间的空间关系进行建模,提取一系列拓扑特征。总而言之,sc-MTOP框架强调了对单个细胞的特征分析,进而可以分析细胞之间的表型多样性及其潜在的临床相关性。
为描绘人类乳腺癌生态系统的全景图谱并探索生态系统学特征的临床相关性,研究团队将sc-MTOP应用于一个具有多组学分析数据的回顾性收集队列637个乳腺癌WSI中,该队列涵盖了所有乳腺癌IHC亚型样本,如HR+HER2-、HR+HER2+、HR-HER2+和TNBC。最终,研究团队共收集了超4.1亿个细胞的原始特征数据,构建了一个空间分辨单细胞数据集。
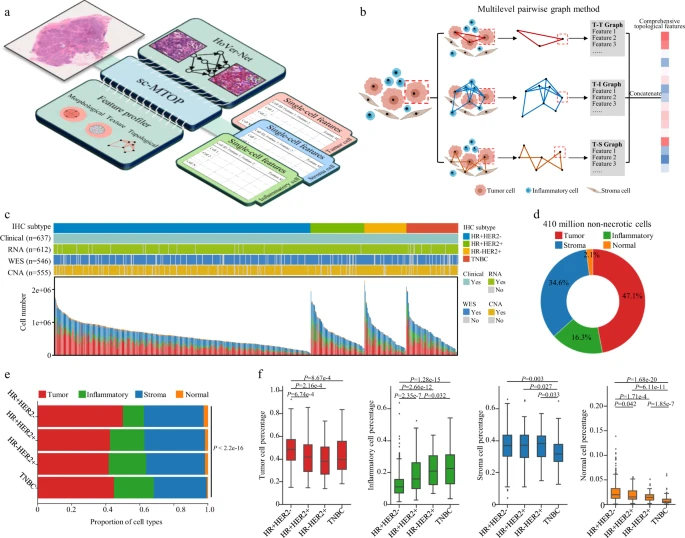
图1. 单细胞形态和拓扑分析以及生成的数据集
基于sc-MTOP数据,研究团队从多个层面剖析乳腺癌生态系统的多样性。首先,在单细胞水平上,通过基于核特征的Leiden聚类分别表征炎症细胞、肿瘤细胞和基质细胞的表型多样性。结果显示,炎症细胞被分为具有不同拓扑特征的10个簇(INF0-INF9):有4个簇(INF0、INF1、INF6和7INF7)由局部聚集的炎症细胞组成,具有高I-I_Nsubgraph、I-I_Degrees和阳性I-I_ClusteringCoefficient的特征,剩余6个簇由空间分散的炎症细胞组成。
研究团队分析了炎症细胞簇与乳腺癌IHC亚型、临床病理特征和患者预后之间的关系,发现不同IHC亚型的乳腺癌具有不同的炎症细胞空间分布模式。I-II级乳腺癌中INF2、INF3、INF4、INF5和INF9细胞较多,而>II级乳腺癌中INF0、INF1和INF6细胞富集。此外,高丰度的INF1和INF7与良好的预后相关。
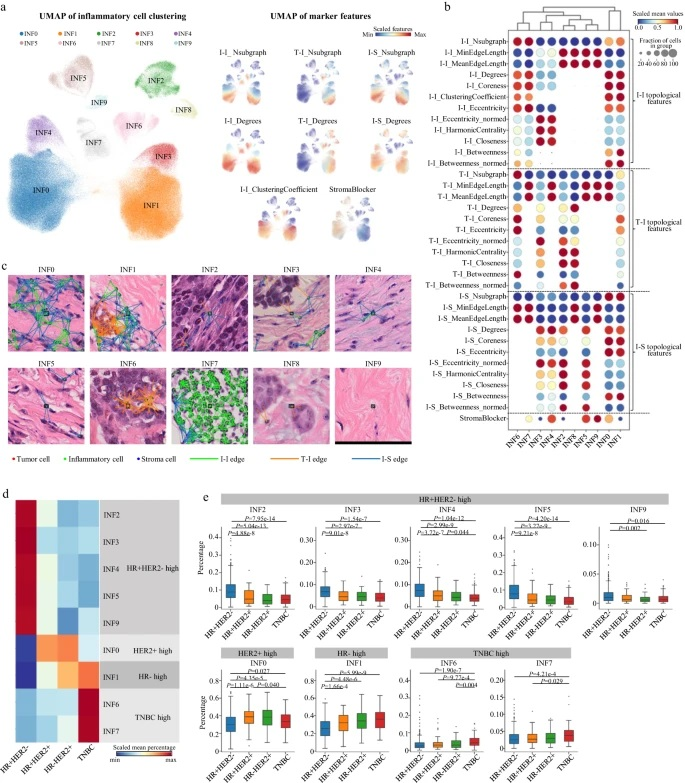
图2. 炎症细胞的单细胞图谱
研究团队将炎症细胞簇的丰度与微环境细胞丰度相关联,探索了炎症细胞簇丰度与抗肿瘤免疫反应之间的关系(图3),发现INF0、INF1、INF6和INF7细胞的丰度与多种免疫细胞亚群的丰度呈显著正相关。此外,这四个簇的丰度也与预测良好免疫治疗反应的生物标志物或基因特征呈正相关。这些结果表明,局部聚集的高丰度炎症细胞与肿瘤免疫微环境的整体激活有关,并可能与乳腺癌免疫治疗的良好反应有关。
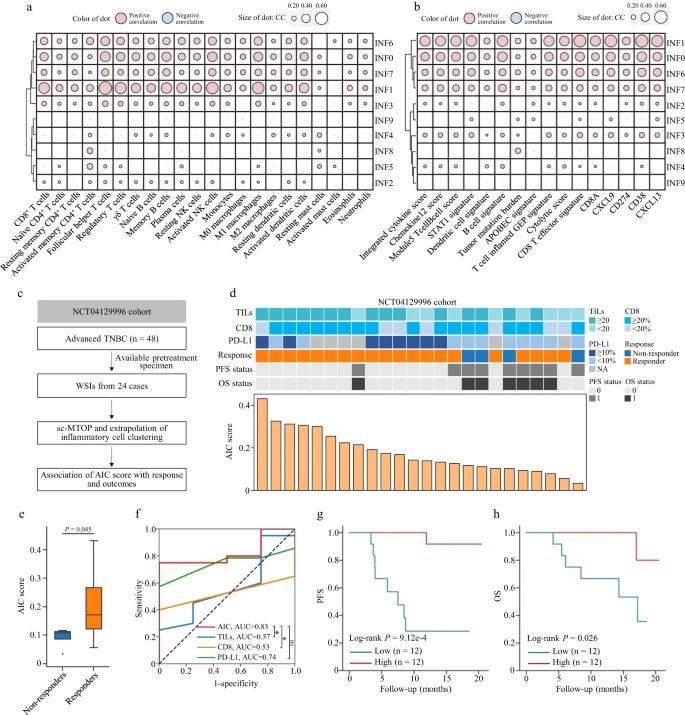
图3. 局部聚集的炎症细胞丰度表明免疫治疗反应
采用相同的聚类分析方法,研究团队分别表征了肿瘤细胞和基质细胞的表型多样性(图4)。
肿瘤细胞被分为12个簇(TUM0-TUM11),具有不同的拓扑和形态学特征;除TUM9和TUM10外,其余簇中肿瘤细胞(98.7%)均呈局部连接,以T-T_Degrees阳性为特征。由与炎症细胞连接的肿瘤细胞组成的4个簇(TUM1、TUM6、TUM7和TUM9)在HR-HER2+和TNBC乳腺癌中富集;由与基质细胞连接的肿瘤细胞组成的2个簇(TUM0和TUM5)在HR+HER2-乳腺癌中富集。高丰度的TUM0和TUM5与不良预后相关。
基质细胞被分为9个簇(STR0-STR8),STR1、STR2、STR4和STR5由与肿瘤细胞有连接的基质细胞组成;STR3、STR6和STR7由基质细胞组成,与肿瘤或炎症细胞均无连接。在> II级乳腺癌中STR0和STR1细胞富集,I-II级乳腺癌中STR2、STR3、STR4、STR6和STR7细胞较多。在预后方面,高丰度的STR2、STR4和STR5与不良预后相关。
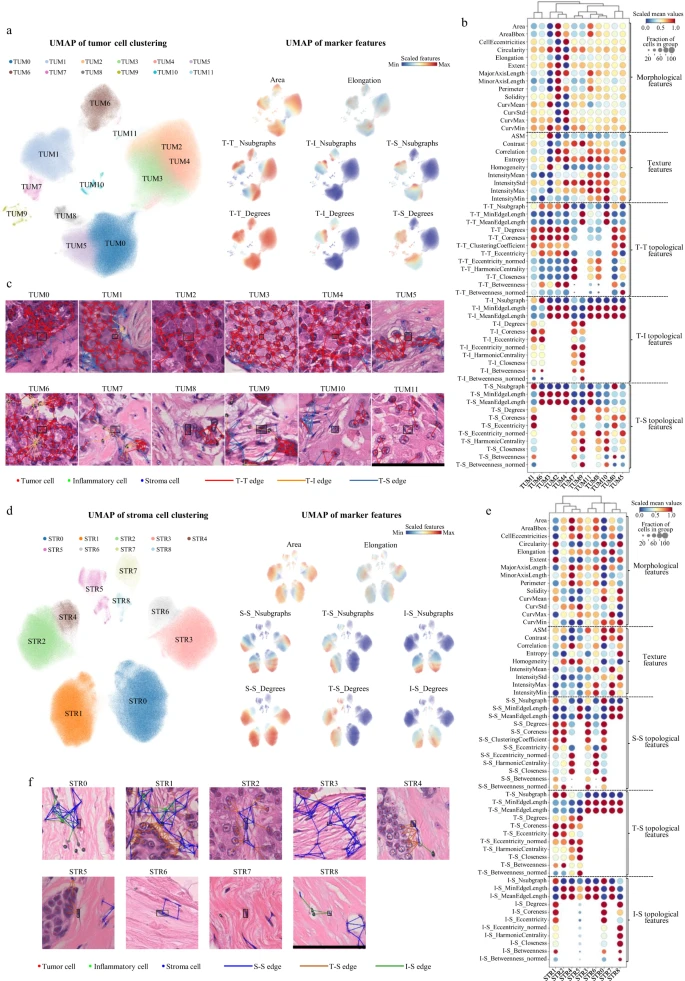
图4. 肿瘤细胞和基质细胞单细胞图谱
肿瘤细胞的无监督聚类鉴定出四个具有相似拓扑特征但不同形态和纹理特征的簇(TUM2、TUM3、TUM4和TUM11),表明肿瘤细胞中细胞核形态具有高度异质性。为此,研究团队量化了每个样本细胞核的形态学肿瘤内异质性(MITH)(图5),发现MITH与肿瘤内遗传异质性之间存在中等相关性。高MITH与侵袭性肿瘤特征相关,包括高T类别、高肿瘤分级、hr阴性和her2阳性状态;TUM4丰度与MITH呈最高正相关。此外,在HR+乳腺癌中,高MITH与细胞周期通路的Cyclin E/ cdk2依赖性激活有关。
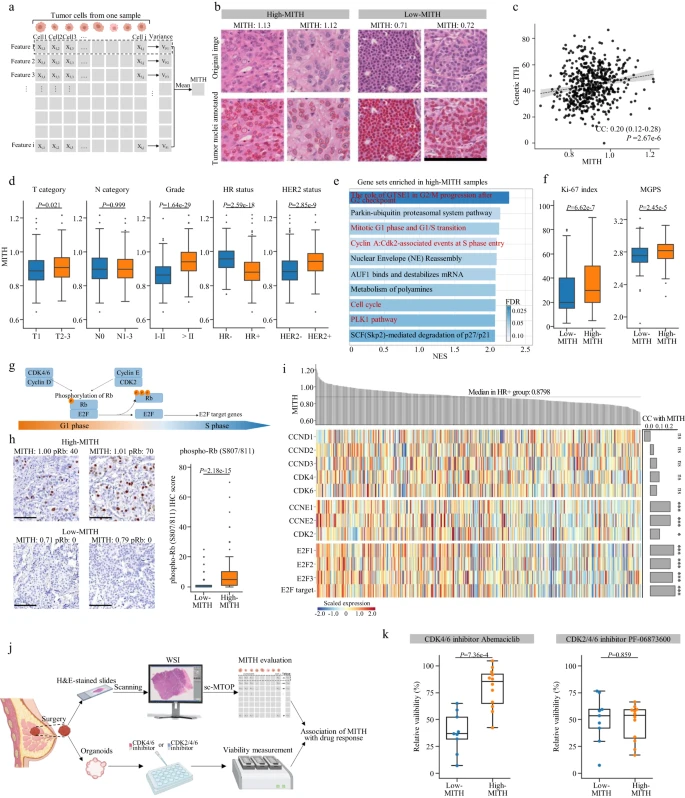
图5. 肿瘤细胞核的肿瘤内形态学异质性表明HR+乳腺癌细胞周期通路活性和CDK抑制剂反应
接下来,研究团队通过空间相关性揭示了细胞簇的共定位,并基于空间相关矩阵的层次聚类定义了微生态模块(MEMs),进一步破译乳腺癌生态系统的空间多样性(图6)。共鉴定出8个MEMs,即Module1_TC、Module2_LT、Module3_DI、Module4_TSModule5_LC、Module6_SI、Module7_TSI和Module8_IA。除Module2_LT和 Module3_DI外,其他MEMs均呈现出有组织的空间模式。与深度学习方法相比,基于sc-MTOP的方法识别的MEMs可以更好地代表肿瘤生态系统中局部多细胞结构的模式,并提供预后相关信息
研究团队对肿瘤MEM成分进行层次聚类,共鉴定出4种生态型(Ecotype 1-4),每种生态型中富集的细胞簇与在该生态型中富集的MEMs一致。进一步比较4种生态型之间肿瘤临床病理特征的差异,发现Ecotype 1和Ecotype 3包含更多淋巴结阳性乳腺癌,Ecotype 2的肿瘤分级较高,4个生态型之间的T分类上没有显著差异。在预后方面,Ecotype 2型乳腺癌患者无复发生存期(RFS)最好,Ecotype 1型乳腺癌患者RFS最差。多因素Cox分析显示,生态型分类是一个独立的预后因素。
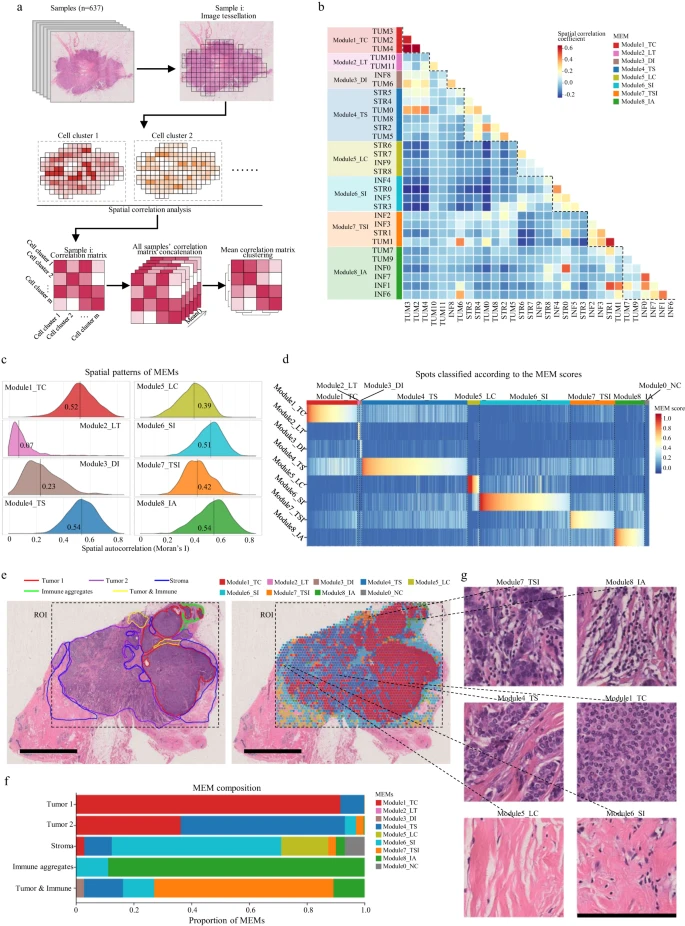
图6. 复发微生态模块表征了乳腺癌生态系统的空间多样性
综上所述,研究团队提出了sc-MTOP,这是一个仅基于常规病理WSI表征肿瘤生态系统的分析框架,并基于该方法系统表征了人类乳腺癌生态系统的表型多样性及其临床相关性。该研究还确定了生态系统特征,可能作为广泛适用的标志物为治疗决策提供信息。sc-MTOP框架和具有匹配临床和多组学数据的大型单细胞数据集可以作为进一步研究的宝贵资源。
参考文献:
Zhao, S., Chen, DP., Fu, T. et al. Single-cell morphological and topological atlas reveals the ecosystem diversity of human breast cancer. Nat Commun 14, 6796 (2023). https://doi.org/10.1038/s41467-023-42504-y
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言














#乳腺癌# #乳腺癌肿瘤# #sc-MTOP#
108